WIPO Sequence ユーザーマニュアル
この文書は、WIPO Sequenceデスクトップアプリケーションを用いた基本的な操作に関する指示をユーザーに提供することを目的としています。通常、ユーザーは、配列表を含む特許出願の提出を検討する、特許出願人またはその代理人です。
このユーザーマニュアルはバージョン3.0.0用です。
1. 機能の概要
次の一覧では、現在のバージョンでツールによって実装されているすべての機能がまとめてあり、関連するセクションへのリンクが提供されています。
| 1つの配列表に関するデータが保存されるプロジェクトを作成する |
| プロジェクトを削除 |
| エクスポートしたプロジェクトから新しく作成したプロジェクトに、データをインポートする |
| ST.25/ST.26配列表ファイルから新規作成したプロジェクトに、データをインポートする |
| XML形式で配列表を検証する |
2. ツールの機能
2.1 プロジェクトホームビュー
このセクションでは、プロジェクトホームで使用できる様々なオプションについて詳しく説明します。
プロジェクトは、配列表の作成に必要なデータを保存するためにツールが使用するオブジェクト構造です。プロジェクトに保存されたデータがWIPO Standard ST.26に準拠していることが確認されると、ツールはこのデータを、作成した配列表内の値として使用します。
このビューには、作成されたプロジェクトのリストが表示され、ユーザーに対し、プロジェクト名、出願人ファイル参照、出願人名、発明の名称、ステータス、または作成日でフィルタリングする検索機能を、並べ替える或いは使用するオプションを提供します。
注: このツールには、最大 1,000 件のプロジェクトが表示されます。プロジェクトがプロジェクトホームビューに表示されない場合でも、ビューに表示されないだけでプロジェクトはローカルに保存されるため、ユーザーは検索機能を使用して名前でプロジェクトを識別してください。
プロジェクトの作成
新規プロジェクトを作成するには、以下のメインのプロジェクトホームビューから開始してください。

1) ビューの上部にある「NEW PROJECT」リンクをクリックします。ここで表示されているように、ツールは「名前」(必須)と「説明」(任意)を要求します。

2) 名前フィールドに値が入力されると、新規プロジェクトを保存できるように、 [Save (保存)] ボタンが有効になります。プロジェクトの一覧では、プロジェクトホームビューに新しいプロジェクトが含まれるようになります。
プロジェクトをインポート
この機能により、以前エクスポートしたプロジェクトをツールにインポートできます。プロジェクトファイルをインポートするには、プロジェクトホームビューから開始する必要があります。
表示されているように、ビューの上部にある[IMPORT PROJECT(プロジェクトをインポート)]のリンクをクリックし、以下の動画の手順に従います。

[Select Range Sequences(範囲配列を選択)] がチェックされていないと、すべての配列がインポートされます。プロジェクトにインポートする配列を選択したい場合は、 [Select Range Sequences] のチェックボックスにチェックを入れ、希望する配列のID番号を適切なフィールドに入力します。単一の配列だけでなく、コンマで区切られた配列のリストやx-y形式の配列の範囲も入力することができます。デフォルトでは、インポートされたプロジェクトの配列の総数が、1〜全ての配列、の範囲として表示されます。
例: 「1, 3, 7, 13-20, 30-50」
プロジェクトが正常にインポートされると、以下の青色のバナーとメッセージがビューの先頭に表示されます。
注:インポートされたzipプロジェクトが現在のデータベースに対応していることを確認する必要があります。プロジェクトが 3.0.0 より前のバージョンでエクスポートされた場合、正しく機能しません。インポートされたプロジェクトは、同じデータベースを使用する必要があります。これは、バージョン 3.0.0 以降の新しいデータベースの実装によるものです。
配列表をインポート
プロジェクトホームビューから、ST.26またはST.25に準拠した配列表からの配列情報だけをインポートできます。それぞれのファイル形式は、ST.26形式では*.xml、ST.25ファイルの場合は*.txtです。手順の詳細については、以下のビデオをご覧ください。

注:配列表をインポートする際、featureとqualifierは大文字と小文字が区別され、WIPO ST.26 のAnnex Iに規定されている値に準拠する必要があります。
また、ST.25に準拠する配列表は、有効である必要があります。そうでない場合、インポートの際、WIPO Sequenceの機能が失われる可能性があります。
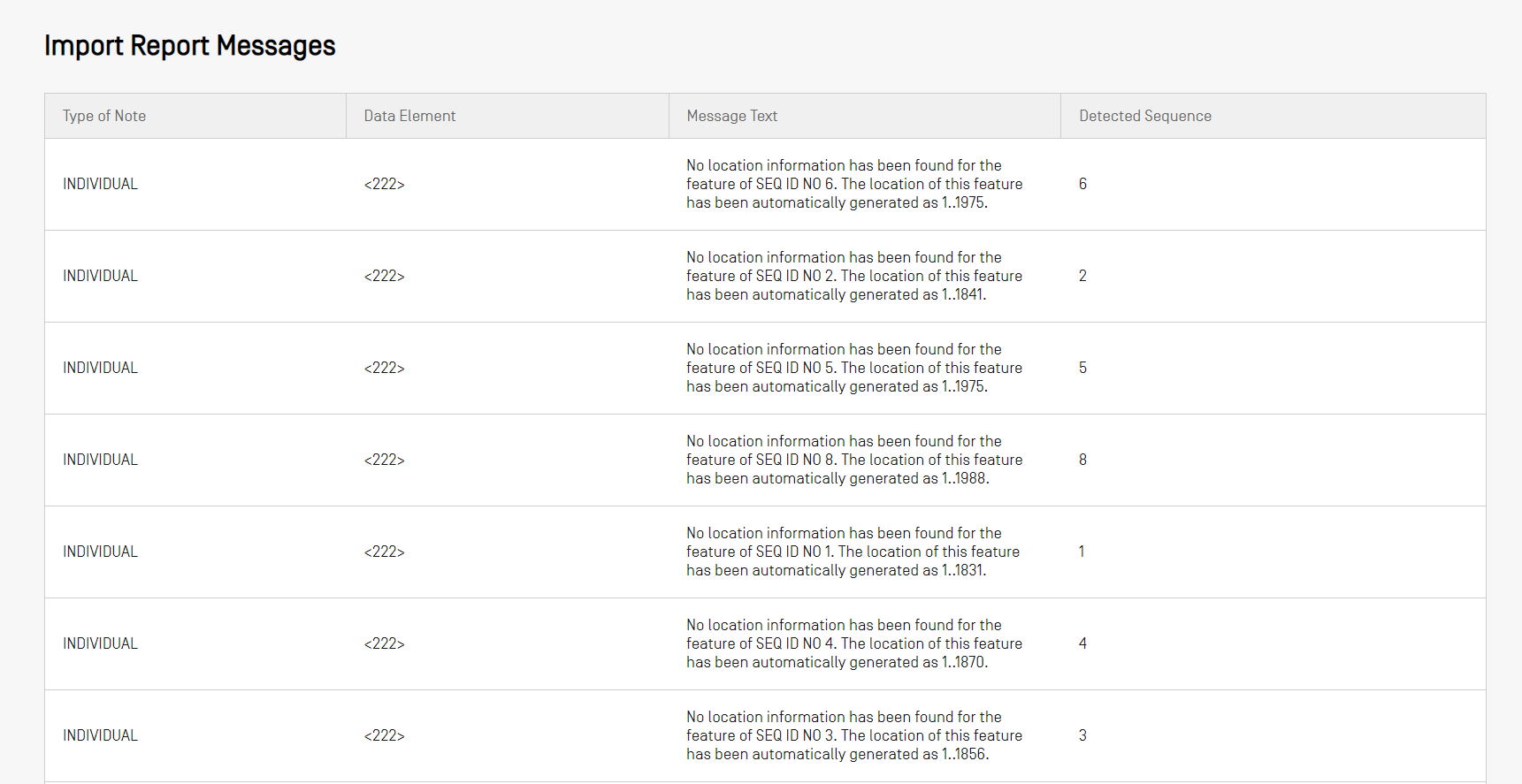
インポートレポートテーブルは、ファイルのインポート時にエラーが発生した場合にのみ表示され、次の列が表示されます。
- 注記のタイプ:特定の配列に関連するメッセージの場合の[INDIVIDUAL(個別)]、または1つ以上の配列全般については[GLOBAL(全般)]。
- データ要素コード:ST.25配列表の場合、ソースファイルに基づく
- メッセージテキスト:特定された問題とその修正のために行われた変更(もしあれば)に関する情報を含む詳細なメッセージ
- 検出された配列:メッセージに関連するインポートされた配列の配列番号(タイプが[INDIVIDUAL(個別)]の場合以外、このフィールドは空白)。
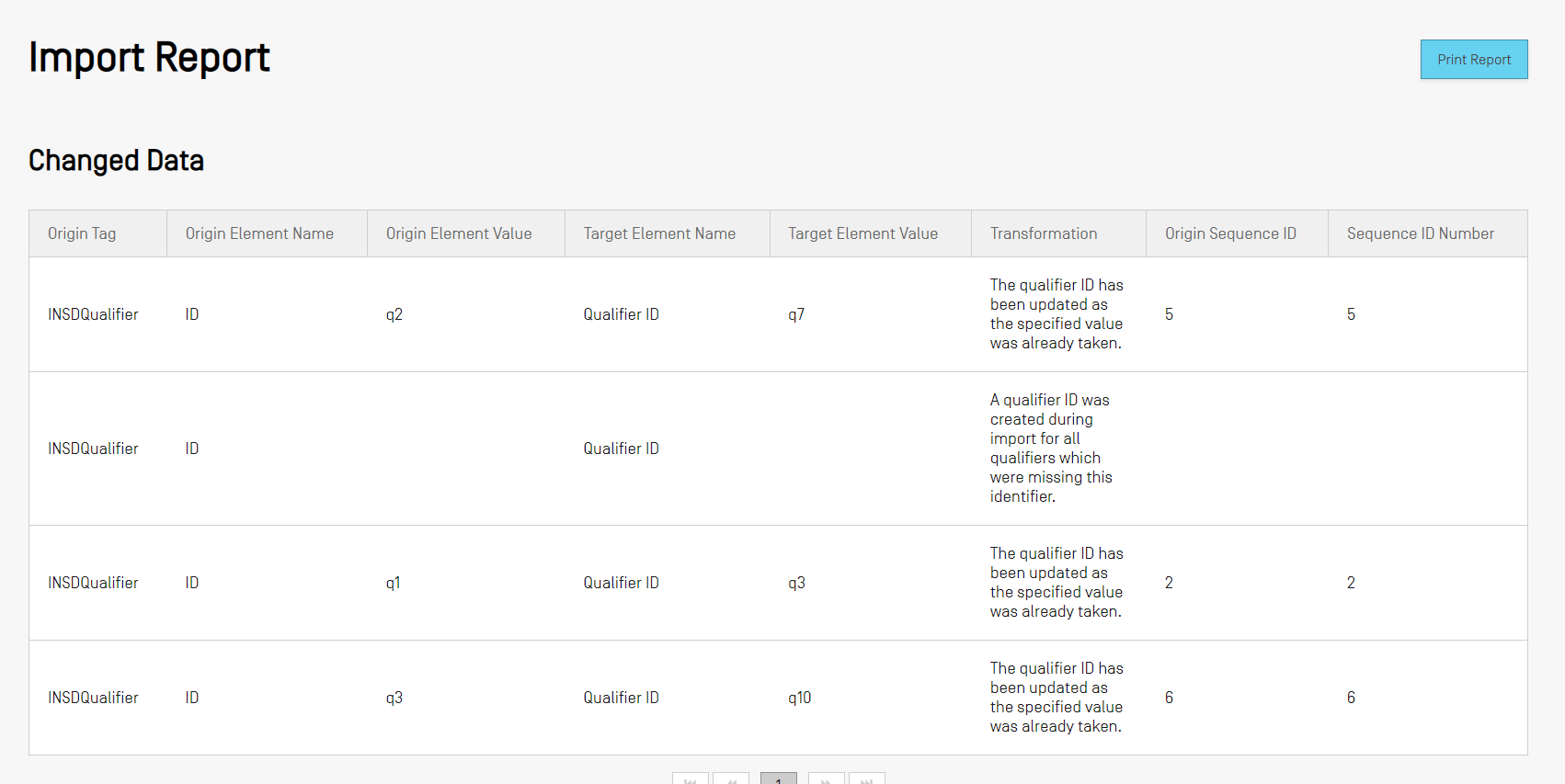
ファイル形式が ST.25 の場合、インポートレポートビューには、最初にインポートレポートと変更データレポートが含まれます。このレポートには、インポートプロセス中に変換または変更されたデータが表示されます。次のデータがサマリーテーブルに示されます。
- 元タグ:WIPO ST.25配列表をインポートする場合の、要素タイプのデータ要素コード
- 元要素名 :要素タイプの対応する名称
- 元要素値 :ソースファイル内の元の要素の対応する値
- ターゲット要素名:プロジェクトに情報を保存する予定の、対応するST.26要素名
- ターゲット要素値:プロジェクトのターゲット要素名に設定される値
- 変換:要素に加えられた変更/変換の説明
- 配列ID番号:プロジェクト内の変換された要素の関連する配列の配列ID番号。
この時点で、プロジェクトホームビューに戻るか、またはこれらの変更のレポートをPDF形式で印刷することができます。PDFファイルをダウンロード/印刷する方法については、 「配列表を表示」の欄を参照してください。
一方、配列表ファイルにエラーがある場合は、インポートプロセスが失敗することがあります。この場合、インポートしようとすると、インポート中にエラーが発生したことを示す赤いバナーが通知されます。

注:このツールは、10万配列のしきい値制限を設けることにより最高のパフォーマンスを発揮します。大規模な配列表を扱う場合、ユーザーは次の対策を実行できます: インポートする配列の特定の範囲を選択し、これらの配列をプロジェクトにおいて範囲ごとにインポートすることによって、インポートプロセスを一連の工程に分割します。例えば、10万配列以下の配列表は、10個×1万配列からなる一連の工程に分割することができ、これらは1つずつインポートすることができます。最初の1万は、プロジェクトの作成に使用されます。
配列表を検証
プロジェクトビューの右上にある [VALIDATE SEQUENCE LISTING(配列表を検証)] ボタンをクリックして、ST.26 配列表ファイルを検証することができます。

配列表が検証を通過すると、バナーが表示されます。

配列表が検証に失敗すると、検証レポートがユーザーのブラウザで開かれ、例示されるように、検証エラーがテーブルに示されます。HTMLファイルの場所は、ユーザーが別の場所にファイルをコピーしたい場合に備えて、XML検証レポートに沿って表示されます。
IEブラウザを使用する場合、形式を正しくロードするためには、お使いのコンピュータで内部スクリプトを実行できるように設定する必要があります。そうでない場合、配列は標準形式で表示されず、読みにくくなります。
配列表を検証するためには、ST.26ファイルが次の要件を満たしていなければなりません。
-
UTF-8でエンコードされていなければならず、XML 1.0仕様に従った有効な文字を含んでいなければなりません
-
次のようにDOCTYPE行を含む必要があります。
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN" "ST26SequenceListing_V1_3.dtd">
ファイルは、DTDファイルST26SequenceListing_V1_3.dtdに準拠していなければなりません。
プロジェクトを削除
プロジェクトを削除するには、プロジェクトホームから開始する必要があります。
削除したいプロジェクトホームテーブル内の行にあるゴミ箱アイコンボタンをクリックします。
図32に示すようにポップアップで[Yes(はい)]をクリックして、選択したプロジェクトを削除することを確認します。

2.2 プロジェクトの詳細ビュー
プロジェクトの詳細ビューには、1つの特許出願または配列表に特有の、すべての情報が含まれています。"一般情報"と"配列データ"の 2 つのセクションに分かれています。上部には、以下を含む、プロジェクトに関する基本情報を含むテーブル があります。
- プロジェクト名
- プロジェクトの作成日時
- プロジェクトに対して最後に行われた更新の日時
- プロジェクトのステータス(可能な値:「新規」/「変更済み」/「作成済み」/「無効」/「有効」/「警告」)- このフィールドは編集不可
- プロジェクトの説明 - 任意
- インポートしたファイルの名前(プロジェクトがインポートされている場合)
- 元のフリーテキスト言語
- 配列数(ラベル:「配列」)
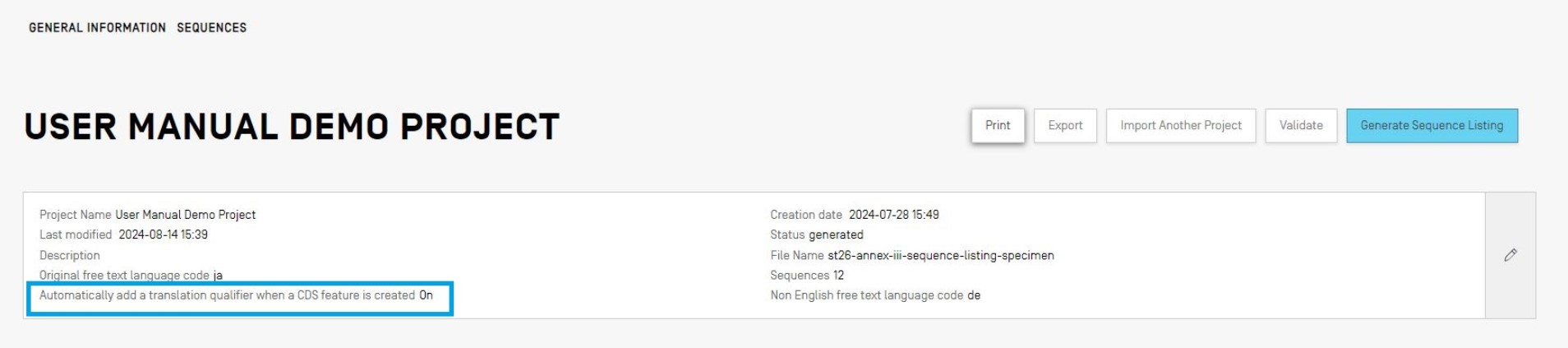
- CDS featureの作成時におけるtranslation qualifierの自動追加を呼び出すためのチェックボックス (プロジェクトレベルの機能)
- 英語以外のフリーテキスト言語
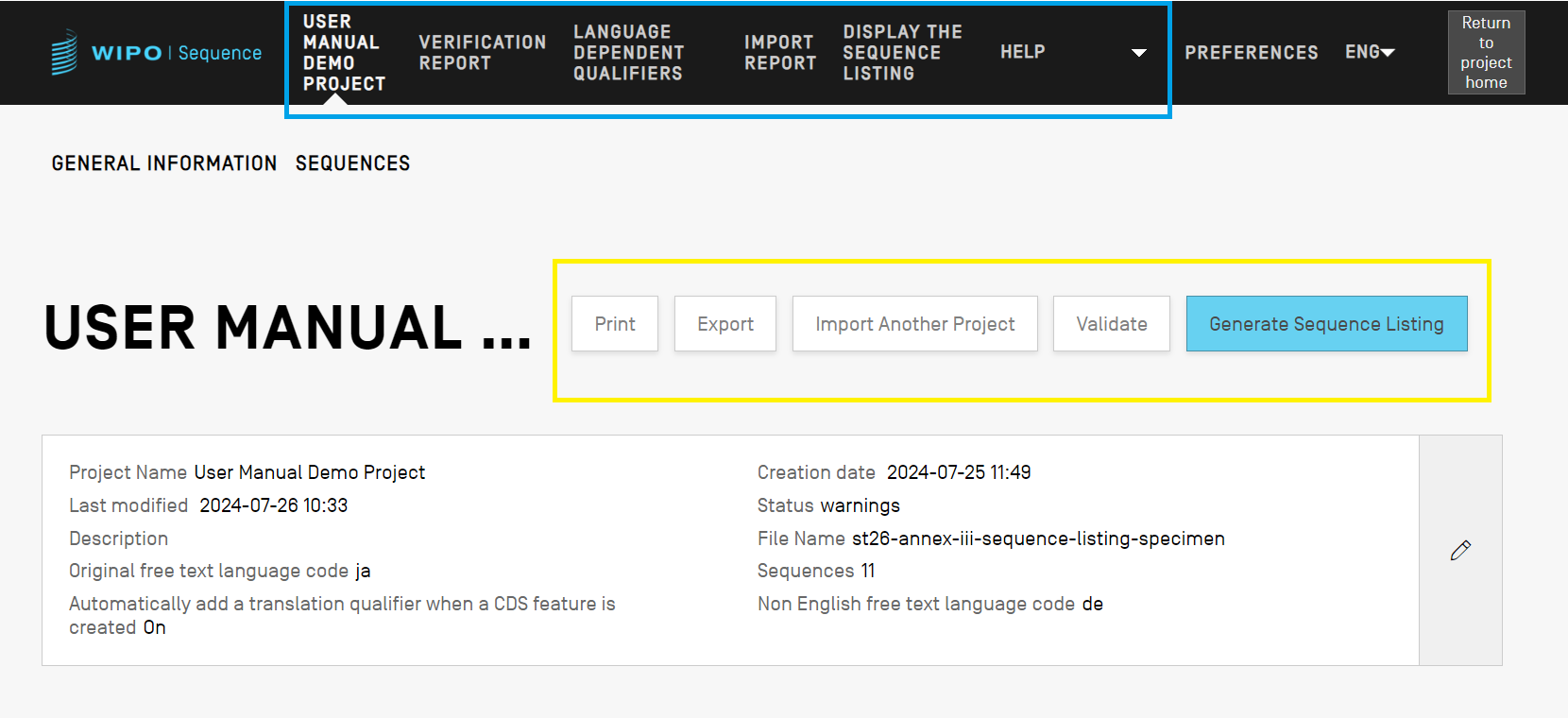
メニューには 2 つのレベルがあり、1 つ目はプロジェクトの詳細ビューのデータに関連するメニュー (黄色で表示)、2 つ目はプロジェクトに関連する他のビューへのナビゲーションメニュー (青色で表示))です。 [Return to project home(プロジェクトホームに戻る)]をクリックすると、プロジェクトから退出できます。
ナビゲーションメニューには、プロジェクトの詳細ページからアクセスできる6つの異なるビューがあります。
- プロジェクトの詳細ビュー(現在のビュー);プロジェクト名で表示
- 検証レポートビュー:ここで検証レポートにアクセスできます
- 言語依存qualifierビュー :ここで言語依存フリーテキストqualifierにアクセスし、エクスポート/インポートできます
- インポートレポートビュー:ここでインポートレポートにアクセスできます
- ディスプレイ配列表ビュー: ここで生成されたST.26配列表の判読可能な形式にアクセスできます
- ヘルプメニュー: ユーザーマニュアル、WIPO SequenceおよびST.26 Knowledge Baseへのリファレンスを含みます
- 環境設定ビュー: WIPO Sequenceのこのインスタンスにおけるすべてのプロジェクトに関係しています
プロジェクトの印刷
プロジェクトを印刷するには、所望のプロジェクトについてプロジェクトの詳細ビューに入り、ビューの先頭にある [Print(印刷)] ボタンをクリックしてください。
次に、プロジェクトから印刷したい情報がどれかを示すための2つのチェックボックス、一般情報および/または配列情報、が表示されます。
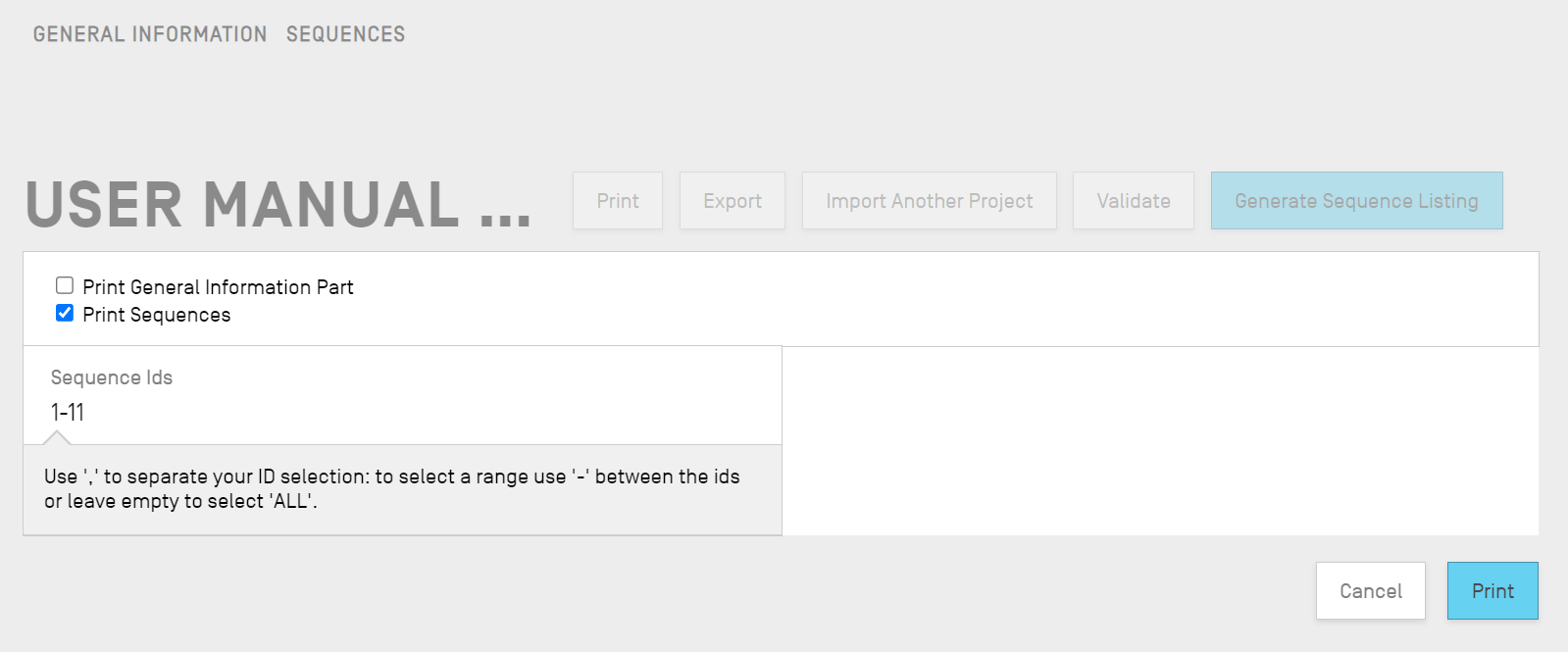
[Print Sequences(配列を印刷)] が選択されている場合、 [Sequence IDs(配列ID)] フィールド内にID番号の範囲を指定して印刷する配列を指定するか、このフィールドを空白のままにしてすべてを印刷するか、選択できます。
デフォルトでは、プロジェクトのシーケンスの合計数が、範囲として表示されます。
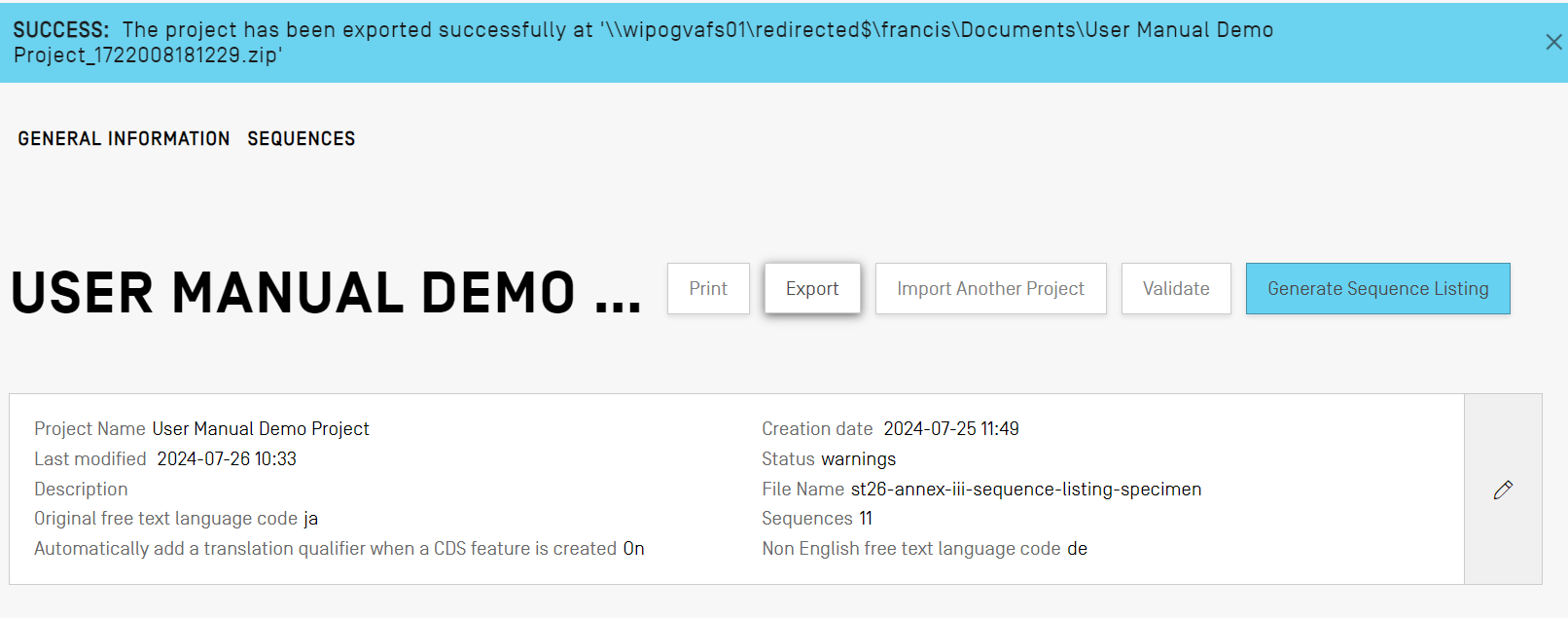
プロジェクトのエクスポート
プロジェクトを、.zipファイルにエクスポートしてプロジェクトデータをバックアップしたり、別のデスクトップコンピュータにインストールされた別のWIPO Sequenceインスタンスにインポートしたりできます。「エクスポート」ボタンをクリックして、.zipファイルを保存する場所を選択するだけです。 エクスポートが成功した場合は、次の青いバナーが表示されます。
現在のプロジェクトに別のプロジェクトをインポートする
ツールに保存されている他のプロジェクトから、現在開いているプロジェクトに情報をコピーできます。このインポートされた情報は、「一般情報」セクションおよび「配列」セクションの一方或いは両方に対するものです。インポートされた一般情報は、プロジェクト内の現在の一般情報と置き換わり、インポートされた配列はプロジェクト内の現在の配列のリストに追加されます。
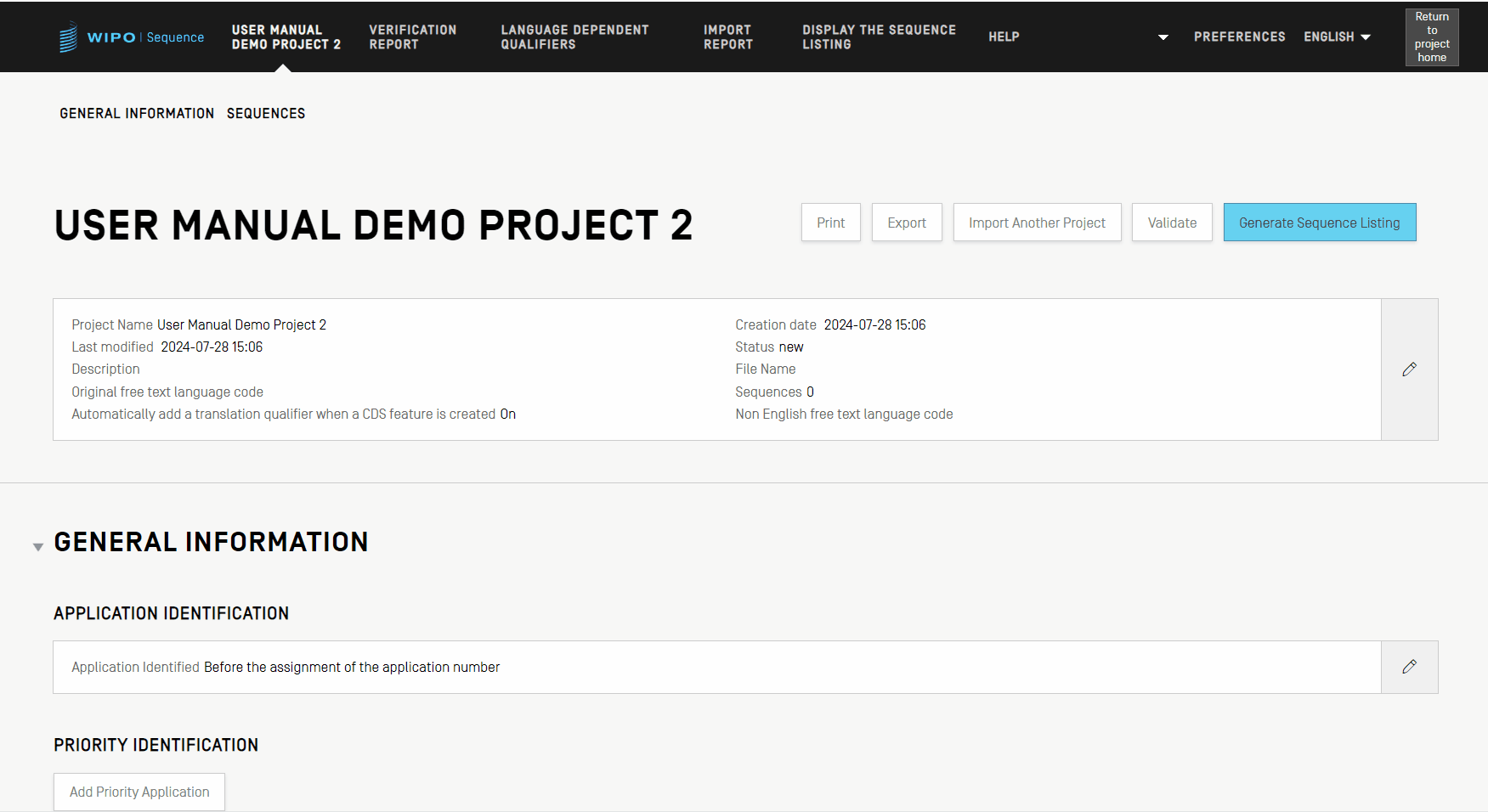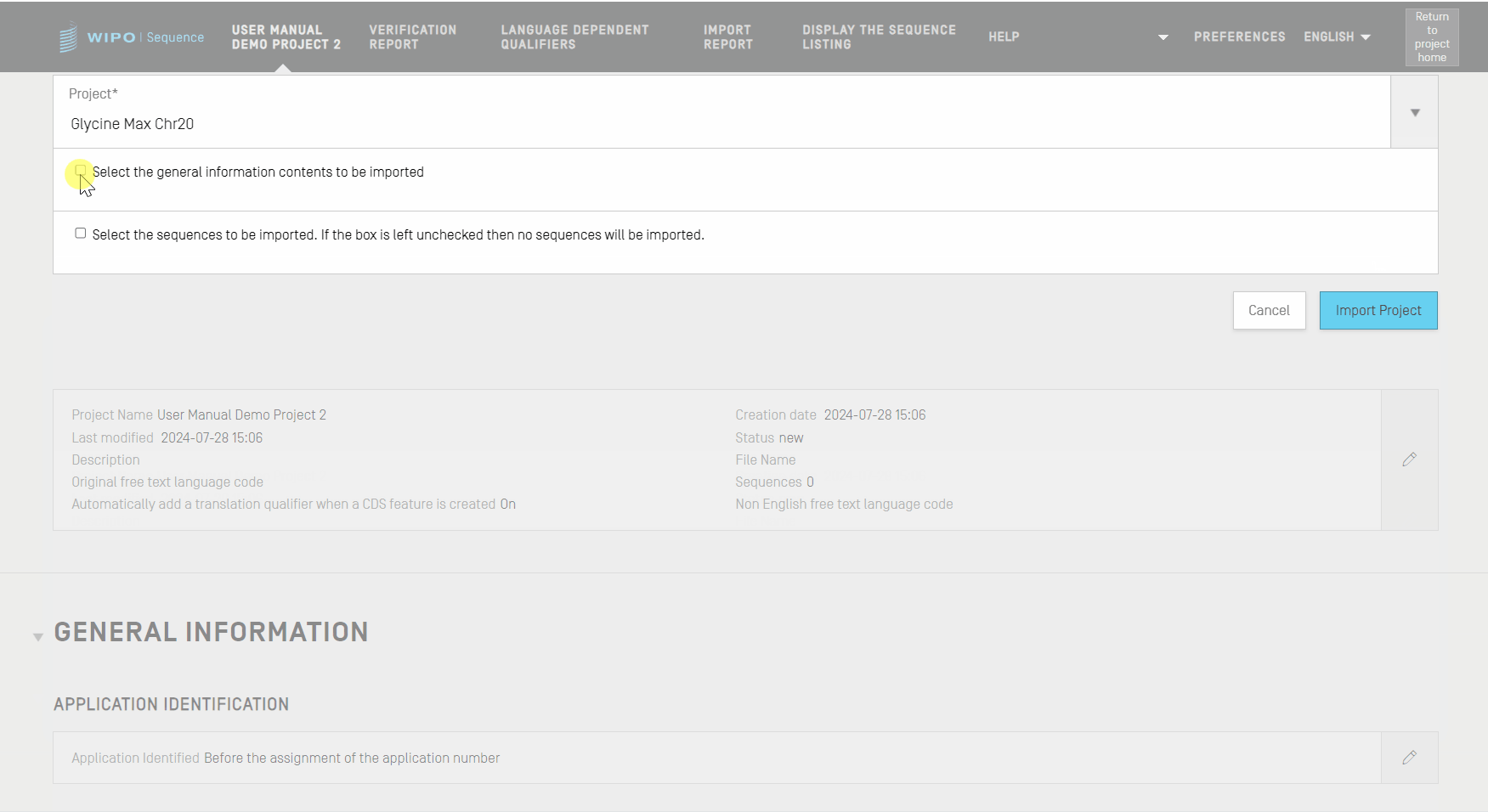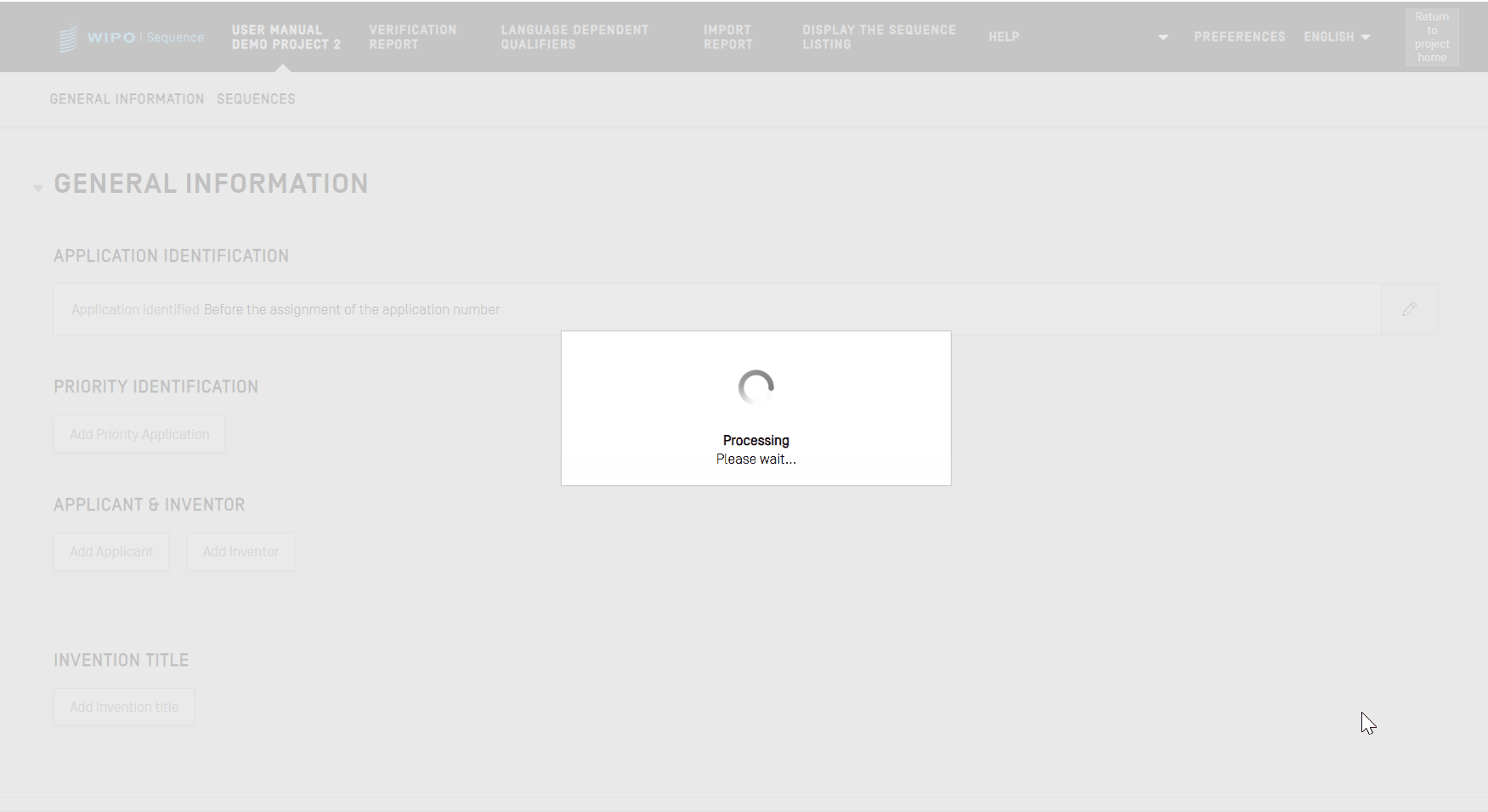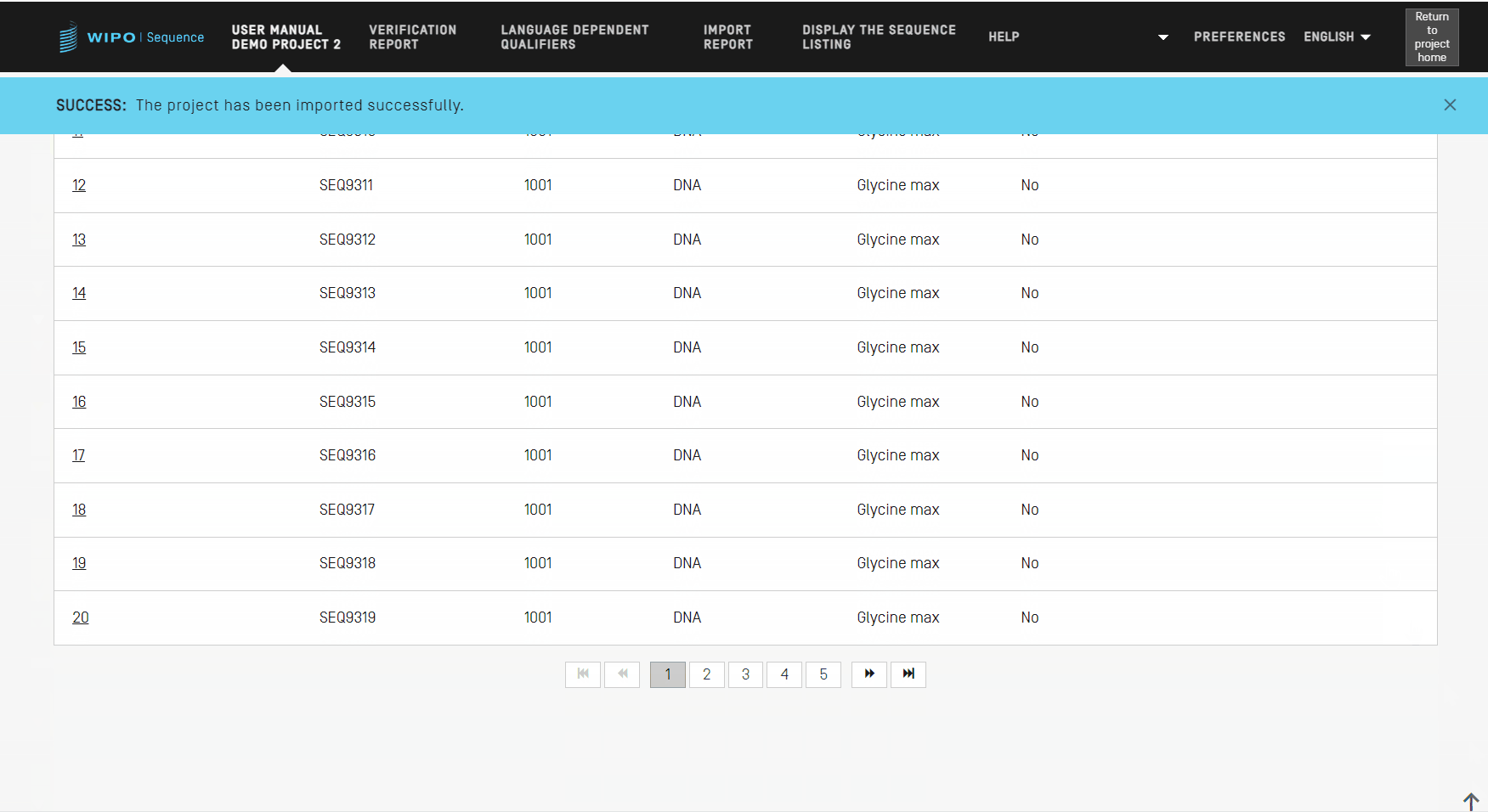
まず、情報をインポートしたいプロジェクトを、表示されているドロップダウンから選択する必要があります。プロジェクトの一般情報セクションに記載されている詳細の一部を含めるかどうか、また、プロジェクトにインポートする配列を指定する配列ID番号の範囲を指定して、配列をインポートするかどうかを選択できます。デフォルトでは、プロジェクトの配列の合計数が、範囲として表示されます。
一般情報チェックボックスがチェックされていると、現在選択されているプロジェクトとインポートされたプロジェクトの両方の一般情報セクションがすべて表示されるテーブルが表示されます。次に、一般情報のどの要素を、インポートされた対応プロジェクトの一般情報で置き換えるか、を選択してください。
最後に、プロジェクトにインポートする一般情報の要素と配列を決定したら、青色の [Import Project(プロジェクトをインポート)] ボタンをクリックしてください。要素が正しくインポートされている場合は、青いバナーが表示されます。
プロジェクトを検証
ST.26準拠のXMLファイルとして配列表を作成する前に、プロジェクトは、あらかじめ検証チェックにかけられます。このステップは、常に、配列表の作成前に実行されますが、単独で実行することもできます。
プロジェクトを検証するには、プロジェクトの詳細ビューの上部にあるValidateボタンをクリックする必要があります。
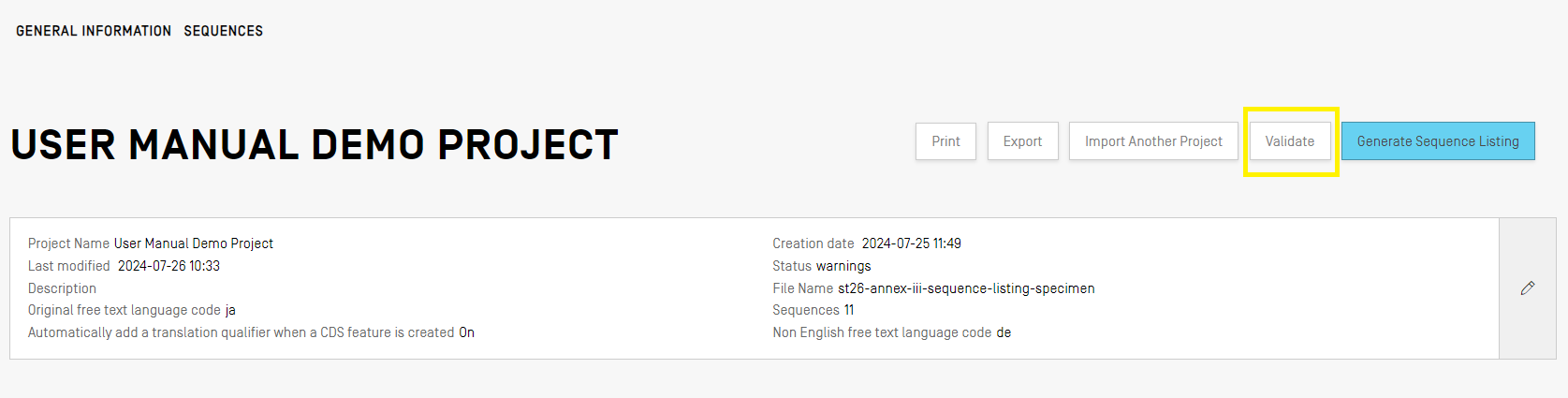
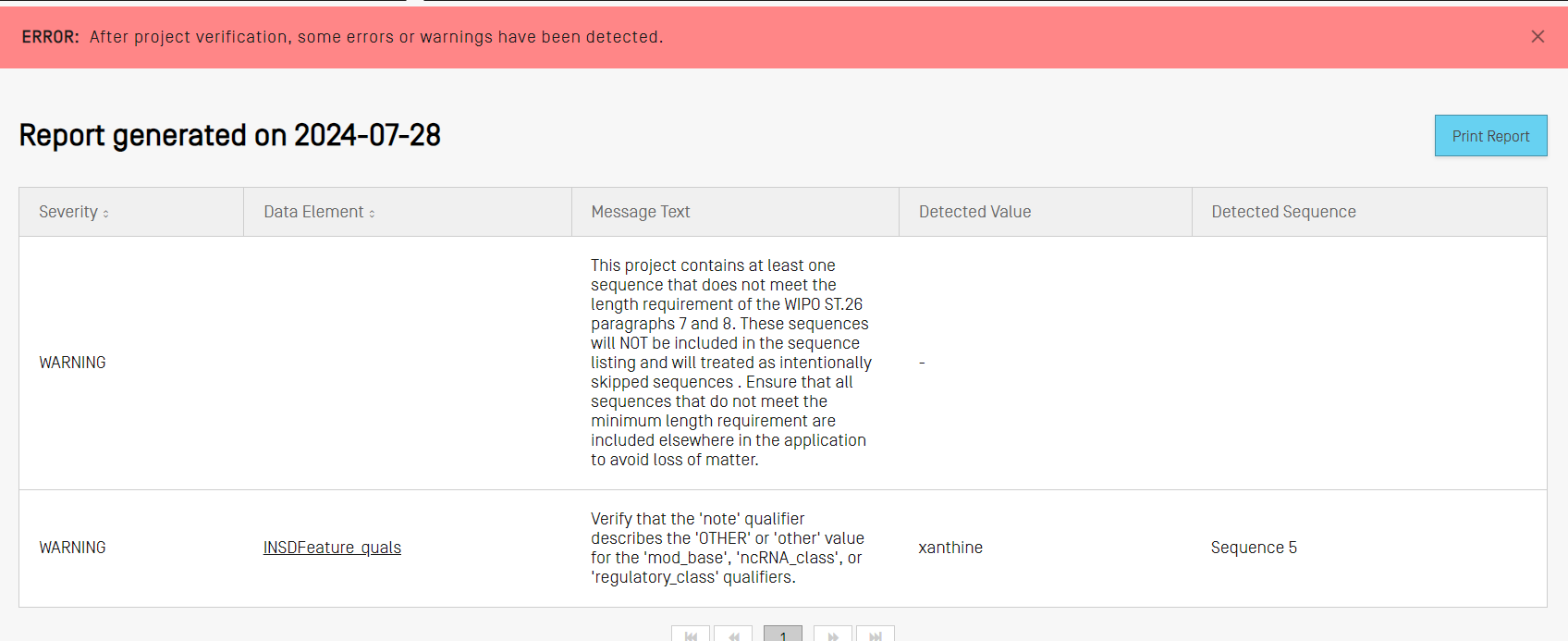
検証が完了すると、ユーザーに[Verification Report(検証レポート)]ビューが表示され、発生し得る検証エラー/警告が表示されます。検証が正常に終了した場合は、青いバナーが表示されます。
検証プロセスでエラーや警告が検出された場合は、検出された検証ルールおよびガイドラインの違反を詳述したテーブルを含む検証レポートが作成されます。各行は、これが対処すべきエラーであるかどうか、または無視できる警告であるかどうかを識別します。
配列表を作成する
プロジェクトに実行できる最後のアクションであり、おそらく最も重要なのは、配列表を作成することです。配列表を作成するには、プロジェクトの詳細ビューの上部にある青色の [Generate Sequence Listing(配列表を作成する)] ボタンをクリックしてください。これにより、まず検証プロセスがプロジェクトに対して実行されます。
プロジェクトが検証プロセスを通過すると、ダイアログボックスが開き、作成したST.26準拠の配列表(.xml)を保存する場所を選択できます。
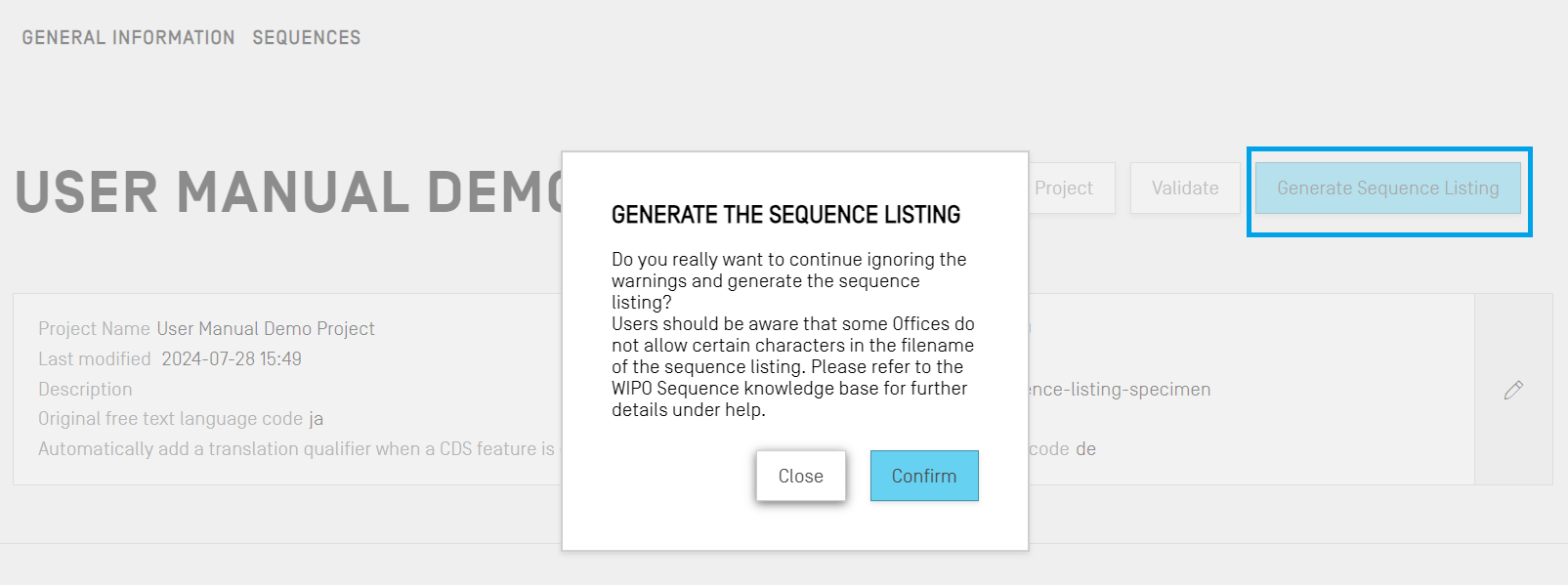
プロジェクトが検証に失敗した場合は、代わりに検証レポートビューが表示され、赤いバナーが表示されます。 プロジェクトが有効な場合は、青いバナーが表示されます。
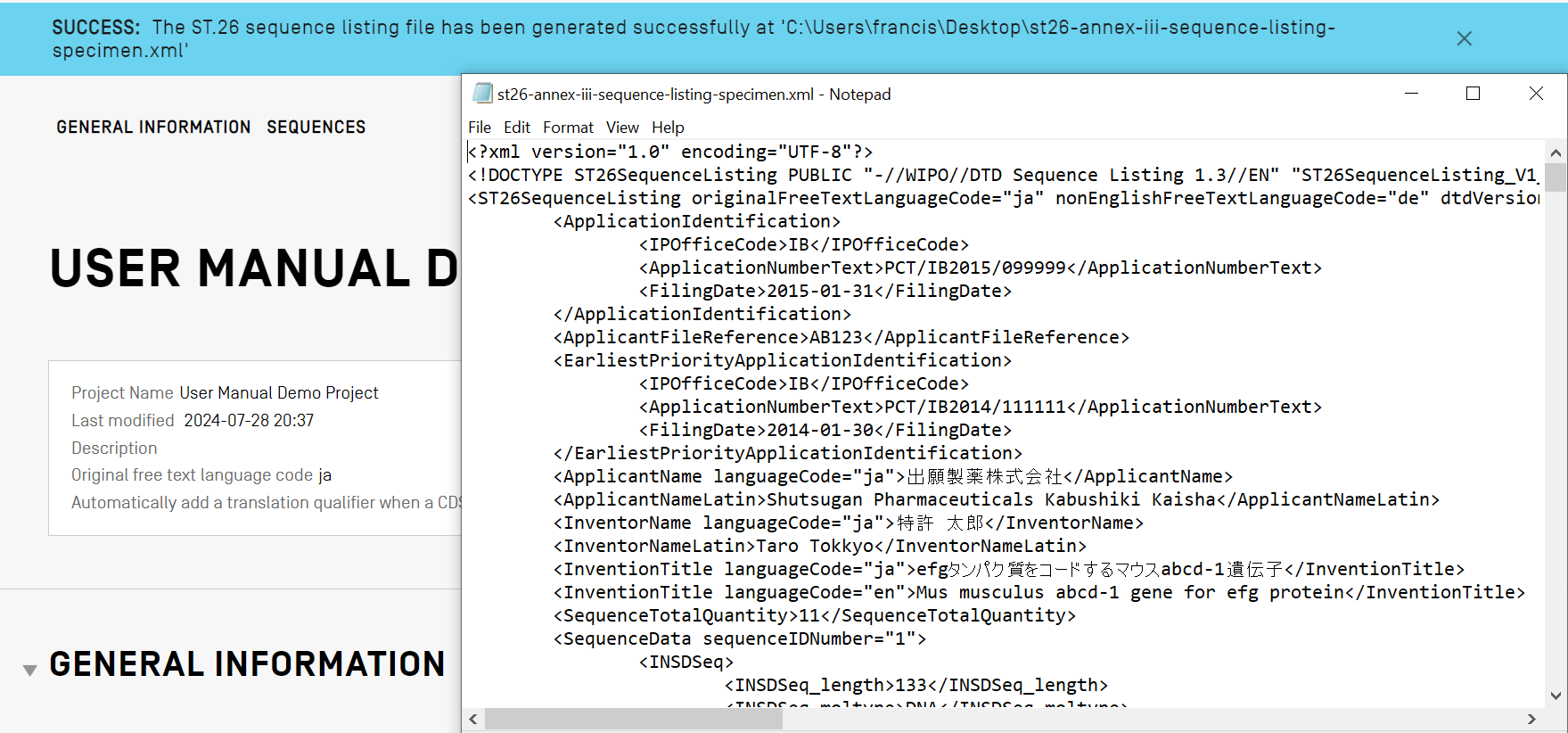
配列表を表示
WIPO Sequenceを使用することで、XMLではなく人が読める形式で配列表を作成できます。配列表が作成された後、XML ファイルを HTML 形式で表示したり、テキストファイルとしてエクスポートしたりできます。任意のインターネットブラウザを使用して、表示されたHTML形式の配列表をPDFファイルとして保存することもできます。エクスポート機能には、[Display the Sequence Listing(配列表を表示)]ビューからアクセスできます。
あるプロジェクトで配列表が正常に作成されなかった場合、Display Sequence Listingビューでは Display Sequence Listing及びExport Sequence Listing as .txt fileのボタンが無効になり、エラーが表示されます
言語依存フリーテキストqualifier
プロジェクトで「フリーテキスト」の値を使用可能なqualifierを、プロジェクトページの「言語依存QUALIFIER」ビュー内でさらに参照します。作業中のプロジェクトに言語依存qualifierを追加すると、このビューにqualifierも表示されます。
qualifierに関連付けられた翻訳フリーテキストの値を変更するには、「Qualifier Name」の値をクリックすると、テーブルの下に編集パネルがあるオーバーレイが開きます。
フリーテキストqualifierをXLIFFファイル形式でエクスポートするには、ソース言語コードとターゲット言語コードを入力する必要があります。 XLIFF ファイルを再インポートする前に、翻訳された値が翻訳者により入力される必要があります。
注:「英語以外のqualifierの値」列に表示される翻訳されたqualifierの値は、英語以外のフリーテキスト言語コードで指定された選択された言語に対応しています。
[IMPORT FREE TEXT QUALIFIERS(フリーテキストQUALIFIERをインポート)] ボタンをクリックすると、ツールがファイルエクスプローラーを開くので、閲覧してインポートしたいXLIFFファイルを探して選択できます。複数の検証ステップを設けることにより、ソースとターゲット言語の値の間で正しいマッピングが実行されていることを確認できます。
XLIFF ファイルからのフリーテキストqualifierのインポート
選択したファイルは、 XLIFF 形式であり、次のデータ項目を含む必要があります。
- プロジェクトの名称
- ターゲット言語コード
- ソース言語コード
- XLIFF単位要素ごとに、以下を含む:
- qualifierの一意なID(形式:文字「q」に続く数字)
- ソース言語タグのqualifierの値
- ターゲット言語タグのqualifierの値
インポート対象として選択したファイルを確認すると、ツールは、一連の検証ステップを確認して続行するかどうかを検証するよう求めます。
- システムは、入力されたファイルのプロジェクト名と選択されたプロジェクトの名前を比較します。
- マッピングできなかったqualifierがあった場合、システムから通知があります。
- システムは、ソース言語とQualifierの値に関連する変更を通知します。
- システムは、ターゲット言語とQualifierの翻訳値に関連する変更を通知します。
これらのステップが完了すると、[SUCCESS: THE FREE TEXT QUALIFIER HAS BEEN IMPORTED SUCCESSFULLY(成功: フリーテキストQUALIFIERは正常にインポートされました)]のバナーが青色で上部に表示され、これとともに、言語依存フリーテキストqualifierに対し以前インポートされた値と現在インポートされた値の詳細を示すインポート・レポートが表示されます。ユーザーは、「RETURN TO FREE TEXT QUALIFIERS」をクリックすると、フリーテキストqualifierビューに戻ることができます。
XLIFF 形式でのフリーテキストqualifierのエクスポート
ユーザーがビューの先頭にある [EXPORT FREE TEXT QUALIFIERS(フリーテキストQUALIFIERをエクスポート)] ボタンをクリックし、ダイアログボックスでqualifierテキストファイルを保存するファイル名と場所を選択すると、プロジェクトのすべてのフリーテキストqualifierがエクスポートされ、XLIFFファイル形式で保存されます。
このファイルには次のものが含まれます:
- プロジェクトソース
- プロジェクトのターゲット言語
- フリーテキストqualifier
- 翻訳されたqualifierのフリーテキスト値
- 表に示した関連するqualifierおよびfeatureの情報
このファイルは、翻訳された適切な値を提供した後、ツールで再度表示、編集、およびインポートできます。
プロジェクトデータの入力
プロジェクトは、作成された配列表と同様、一般情報および配列データの2つのセクションに分割されます。データをプロジェクトにカットアンドペーストしたり、別のプロジェクトや配列表からデータをインポートしたりできます。通常、鉛筆アイコンをクリックすると、プロジェクトフィールドを編集できるようになります。必須フィールドはアスタリスク(*)で示されます。
一般情報
一般情報のセクションでは、特許出願自体に関連する情報を入力できます。この情報は作成した配列表を本出願に関連付けるために使用されます。最初のサブセクションである出願の識別サブセクションは、選択したプロジェクトの特許出願のステータスおよび情報に関連しています。以下では、このセクションで提供する必要がある情報を順を追って説明します。
ステップ 1: 出願の識別
出願の識別サブセクション内の情報を編集するには、サブセクションの右側にある強調表示された鉛筆アイコンをクリックします。次に、以下の手順に基づいて情報を入力してください。
- 出願に出願番号がすでに割り当てられている場合、出願した知的財産庁(特許庁)のコードを選択してください。これはWIPO Standard ST.3 コードです。
- 出願番号をすでに受け取っているか、または出願人ファイル参照番号を提供しているかを、適切なラジオボタンを選択して特定する必要があります。
- 出願番号がない場合、このフィールドに出願人ファイル参照番号を入力しなければなりません。
- 出願番号がすでに割り当てられている場合は、付与された出願番号を入力する必要があります。
- 日付が割り当てられている場合は、日付ピッカーで出願日を選択します。
- 青色の [Save(保存)] ボタンをクリックします。
ここに示す例では、すべてのオプション値も入力されています。

注: 必須の値が入力されている場合でも、検証レポートには常に「出願識別番号がありません。出願番号が割り当てられている場合、出願番号は必須です。」という警告が表示されます。
ステップ 2:優先権出願
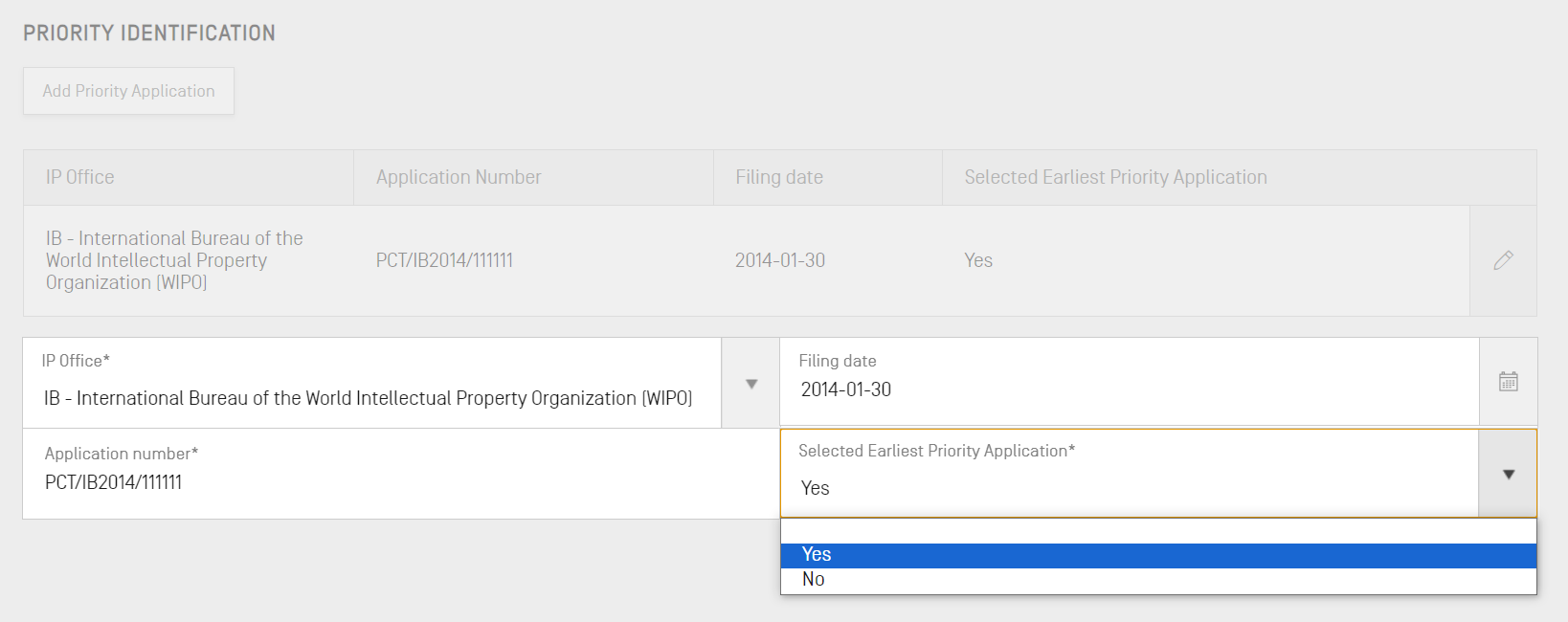
次に、本出願に関連する優先権出願がある場合、これらの詳細を優先権出願サブセクションに入力します。優先権出願をプロジェクトに追加するには、一般情報セクションにある [Add Priority Application(優先権出願を追加する)] ボタンをクリックしてください。
現在選択されている優先権出願を最先として設定するには、 [Selected Earliest Priority Application(選択されている最先の優先権出願)] ドロップダウンで [Yes(はい)] を選択してください。これにより、配列表の作成時に、最先の優先権出願として設定または変更されます。終了するには、青色の [Add Earliest Priority Application(最先の優先権出願を追加)] ボタンをクリックします。
ステップ 3:出願人/発明者
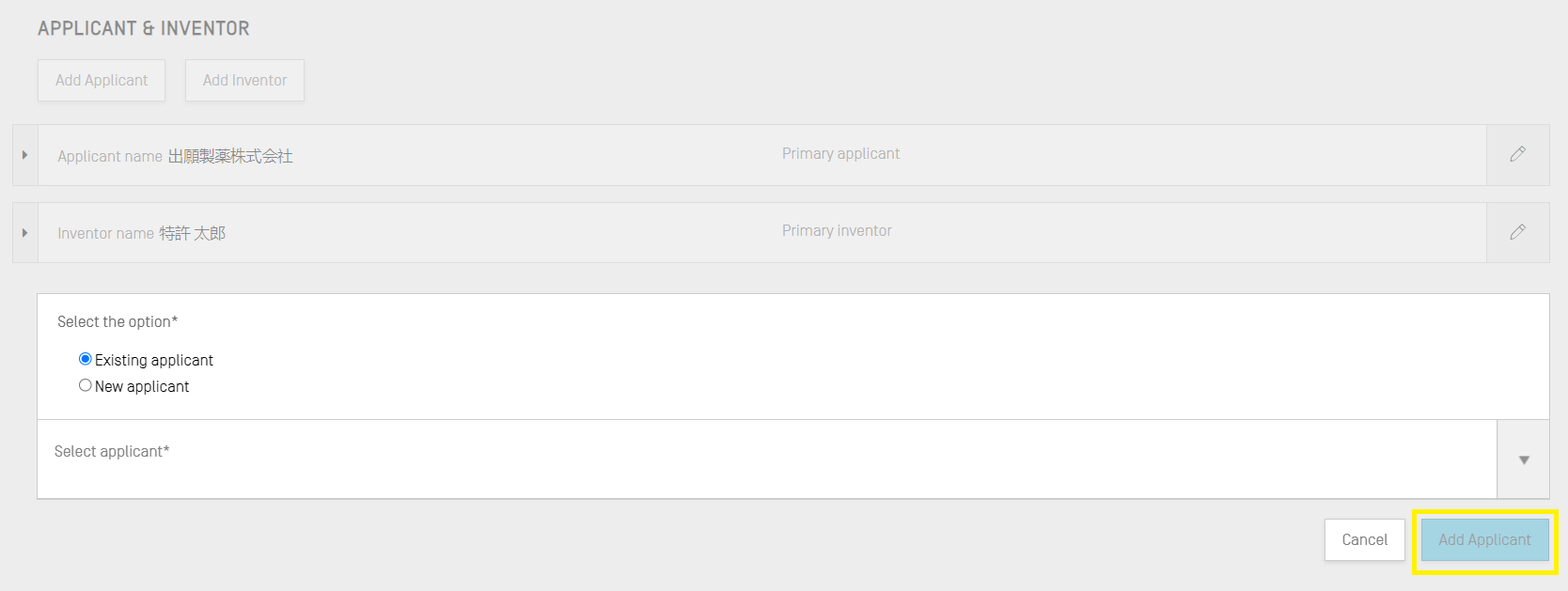
新規出願人または発明者に関するデータをプロジェクトに追加するには、プロジェクトの詳細ビューの一般情報セクションにある [Add Inventor(発明者を追加)] または [Add Applicant(出願人を追加)] ボタンをクリックしてください。これらの作業を実行するための手順は同じであり、ここでは一般的な説明のみとしますが、出願人と発明者の両方をプロジェクトに含める場合は、出願人が発明者でもある場合であっても、このプロセスを2回繰り返す必要があります。
ボタンをクリックすると、オーバーレイが開き、2つのラジオボタンが表示されます。[Existing applicant/inventor(既存の出願人/発明者)] が選択されている場合は、デスクトップツールのローカルインスタンス内に現在保存されている個人および法人の一覧を表示するドロップダウンボックスから選択できます。
[New applicant/inventor(新規出願人/発明者)] が選択されている場合、新規個人/法人を作成するときと同様に詳細を入力してください。
詳細の入力が完了したら、 [Add Applicant/Inventor(出願人/発明者を追加)] ボタンをクリックしてください。
注:配列表が有効と見なされるために必要な出願人は 1 人だけです。よって、1人の出願人および/または発明者をメインとしてマークする必要があります。これは、生成される配列表に表示される出願人/発明者です。
ステップ 4:発明の名称
最後のステップは、一般情報セクション内に[Invention Title(発明の名称)]を追加することです。
新しい発明の名称を追加するには:
- [Add Invention title(発明の名称を追加)] ボタンをクリックします。
- 表示されるオーバーレイで、発明の名称を入力し、発明の名称が記載されている言語も示してください。
- 青色の [Add Invention title(発明の名称を追加)] ボタンをクリックします。
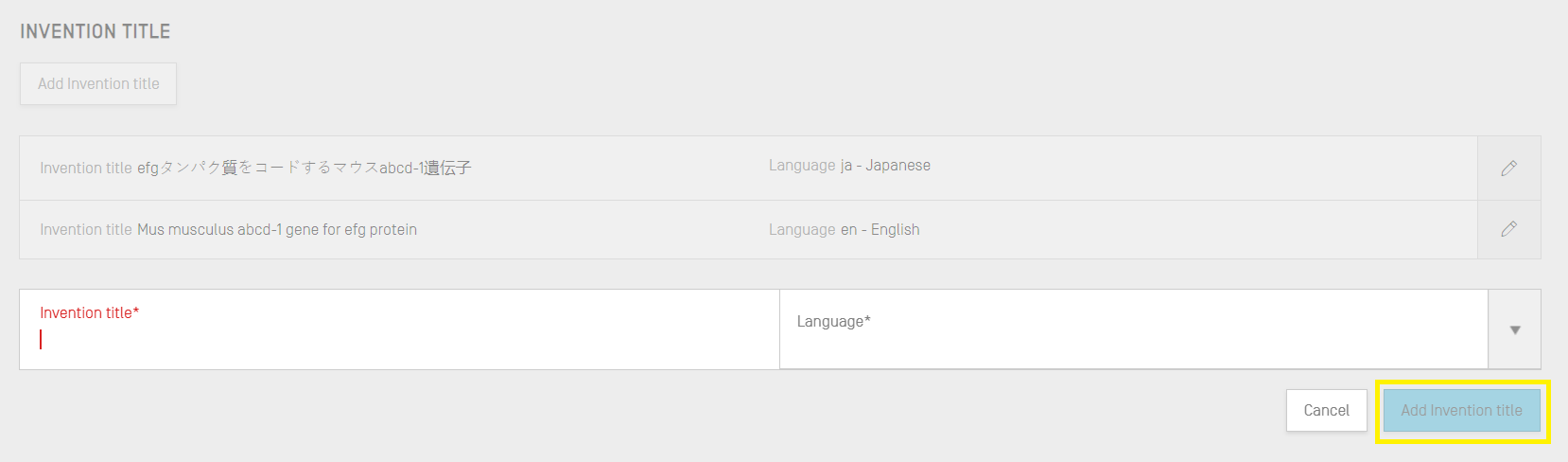
WIPO ST.26では、配列表において発明の名称は出願した言語で記載されなければなりません。ただし、必要に応じて、プロジェクトは別の言語で複数の発明の名称を含むこともできますが、言語ごとに1つの発明の名称のみ可能です。 新しい発明の名称は、上記の手順で追加することができます。
配列データ
プロジェクトの詳細ビューの「配列」セクションでは、配列自体に関連する技術情報を入力します。WIPO Sequenceは、配列の手動作成、インポート、挿入など、プロジェクト内で配列データを入力するためのさまざまな手段を提供します。以下のサブセクションでは、これらの作業を実行するために必要な手順について詳しく説明します。
配列を作成
プロジェクトで配列を作成する手順は次のとおりです。
- [Create new sequence(新規配列を作成)] ボタンをクリック。[Sequence Panel(配列パネル)]が表示されます。
- [Sequence Name(配列名)]のフィールドに希望する配列名を入力して、配列名を特定して下さい。あるいは、[Sequence Name]のフィールドを空白のままにして下さい。その場合、WIPO Sequenceが新規配列にデフォルトの名を割り当てます。デフォルト名は、「Seq」および反復的に増加する数からなります(「Seq_1」、「Seq_2」、「Seq_3」)。
注: [Sequence Name]は、ユーザーがプロジェクト内の各配列を区別しやすくするために設けられており、ここでの配列名は、配列表 XML ファイルには表示されません。
- [Molecule Type(分子型)*]のドロップダウンボックスを用いて、WIPO ST.26で許可されている3つの分子型(「DNA」、「RNA」、「AA」)のいずれかを選択して下さい。「DNA」および「RNA」の両方のセグメントを含む配列を作成したい場合は、「DNA」を「Molecule Type」として選択してください。
- 配列残基を[Residues(残基)*]のフィールドに入力または貼り付けます。
- [Organism name(生物名)*]のフィールドにソースとなる生物名を、入力するか貼り付けて下さい。事前に定義された生物名のリストから生物名を選択する場合、生物名を入力し始めると、ドロップダウンリストが表示されます。希望する生物名がある場合は、それを選択します。そうでない場合、ローカルに保存されているユーザーの生物データベースに生物を追加するよう、ツールから求められる場合があります。
- 分子型が「DNA」または「RNA」の場合は、[Qualifier Molecule Type(Qualifier分子型)]のドロップダウンボックスから特定の分子型を選択する必要があります。これには、ステップ3で選択した分子型に適した値が入力されます。なお、分子型が「AA」の場合、[Qualifier Molecule Type]には「protein」の値が自動的に入力されます。
- [Mark as an intentionally skipped sequence(意図的にスキップされた配列としてマークする)]のチェックボックスがチェックされている場合、その配列は、[intentionally skipped sequence(意図的にスキップされた配列)]として配列表XMLファイルに含まれます。つまり、WIPO ST.26、 paragraph 58で指定された形式になります。[Mark as an intentionally skipped sequence]ボックスは、次の配列を保存するためにチェックする必要があります:非nヌクレオチド残基が10個未満の配列;非Xアミノ酸残基が4個未満の配列。配列が意図的にスキップされた場合、配列パネルでは必須要素の値の入力に対するすべての制約が解除されます。その結果として保存された配列は、プロジェクトを検証する際、無視されます。
- 配列の分子型が「DNA」の場合、[The sequence contains both DNA & RNA fragments(配列がDNAとRNA両方のフラグメントを含んでいます)]とのラベルを有するボックスが表示されます。配列にDNAフラグメントとRNAフラグメントの両方が含まれている場合、このボックスにチェックを入れると、WIPO ST.26、paragraph 55でDNA/RNAハイブリッドを記述するために必要なfeatureを簡単に追加できるようになります。[The sequence contains both DNA & RNA fragments]のボックスにチェックを入れると、配列パネルが拡張され、「misc_feature」のfeature keyと対応する位置で各DNAおよびRNAフラグメントを記述できるフィールドが表示されます。各フラグメントについて、Molecule Typeのフィールドのドロップダウンメニューから「DNA」または「RNA」を選択し、Locationのフィールドに位置を入力します。必要に応じて、追加の文言を「Further Text(追加のテキスト)」のフィールドに含めることができます。ユーザーは、[Add new ‘misc_feature’ feature(新規「misc_feature」featureを追加)]のボタンをクリックして、必要な数だけこれらのfeatureを作成できます。なお、配列の全長は、各残基が「DNA」または「RNA」として示されるように、「misc_feature」のfeature keyでカバーする必要があります。
- 終了して配列を保存するには、グレーの[Create sequence(配列を作成)] ボタンまたは青色の [Create & Display Sequence(配列を作成して表示)] ボタンをクリックします。青い[Create & Display sequence]ボタンをクリックすると、配列の作成後に折りたたみ可能な配列の表示が開き、値を確認できます。これは、プロジェクトの詳細ビューの配列セクションの下に表示されます。
注: 配列がヌクレオチド配列(「DNA」または「RNA」)の場合、WIPO ST.26、Annex I、Table 1に記載されている記号(a、c、g、t、m、r、w、s、y、k、v、h、d、b、n)のみが許可されます。記号「u」は許可されません。大文字で入力された記号は小文字に変換されます。文字以外の入力記号(スペース、数字、*、-など)は全て自動的に削除されます。
配列がアミノ酸配列(「AA」)の場合、WIPO ST.26、Annex I、Table 3に記載されている記号(A、R、N、D、C、Q、E、G、H、I、L、K、M、F、P、O、S、U、T、W、Y、V、B、Z、J、X)のみが許可されます。アミノ酸記号は1文字で入力する必要があります。アミノ酸配列が 3 文字のアミノ酸記号 (Met-Arg-Leu-Trp-Ile など) で表される場合は、まず 1 文字の記号に変換してから、[Residues]のフィールドに入力または貼り付ける必要がありますp>
新しく作成された配列は、配列のリストの最後にあり、次に使用可能な配列ID番号が付与されています。リスト内の配列の位置を並べ替えるには、 この手順に従います。
配列をインポート
配列をファイルからプロジェクトに直接インポートすることもできます。使用可能なファイル形式のリストはsection 3に示されています。入力ファイルを選択すると、デスクトップツールがファイルで使用されている形式を自動的に検出します。
- 開始するには、[Import sequence(配列をインポート)] のボタンをクリックしてください。
- [Upload file [.txt, .xml](ファイル [.txt, .xml]をアップロード)] をクリックしてください。ダイアログボックスが開いたら、インポートする配列データを含むファイルを選択します。デスクトップツールは、使用されている形式を検出し、インポート時にいくつかの検証チェックを実行します。このツールでは、プロジェクトへの配列のインポートに5つの形式(ST.25、ST.26、マルチシーケンス、RAW、FASTA)が使用できます。
- WIPO ST.25またはST.26形式のファイルを選択した場合、最初に[Select Range Sequences(範囲配列を選択)] のチェックボックスが表示されます。 チェックすると、ファイル内の各配列の配列ID番号とプロジェクトで提供される配列のリストに配列が追加される順序を含むテーブルが開きます。すべての配列をプロジェクトにインポートしたくない場合は、配列ID番号の所望の範囲を指定できます。単一の配列だけでなく、コンマで区切られた配列のリストやx-y形式の配列の範囲も入力することができます。たとえば、「1、3、7、13-20、30-50」などです。
- マルチシーケンス形式のファイルをインポートする場合、 [Select Range Sequence IDs(範囲配列IDを選択)] のチェックボックスが表示されます。これをチェックすると、ファイル内の対応する配列の配列ID番号、および各配列の詳細(配列名、分子型、生物名を含む)を示すプレビューテーブルが表示されます。プロジェクト内の配列のリストにインポートしたい配列ID番号の範囲を選択してください。デフォルトでは、選択された配列表ファイルの配列の合計数が、範囲として表示されます。
- 配列のインポートプロセスで使用可能な最後の2つの形式は、RAWおよびFASTAファイル形式です。これらの形式は、ファイルごとに1つの配列だけを定義します。インポート用にRAWファイルまたはFASTAファイルを選択すると、ツールは関連するパネルを表示します。必須フィールドを入力して、続行してください。
- インポートが成功すると、ツールは[Import Report(インポートレポート)]ビューに移動します。
配列を挿入
配列のリストの特定の位置に配列を挿入するには、[Insert Sequence(配列を挿入)] ボタンをクリックしてください。すると、オーバーレイパネルが表示されます。配列の作成に必要なすべての情報を入力するだけでなく、パネルの左上で、配列のリストにおいて配列を表示する位置を入力する必要があります。終了するには、 [Insert sequence(配列を挿入)] または [Insert & Display Sequence(配列を挿入して表示)] をクリックします。

配列を並べ替える
以下の動画に示すステップを用いて、プロジェクトで提供されている配列表内で配列を表示する順序を再編成できます。

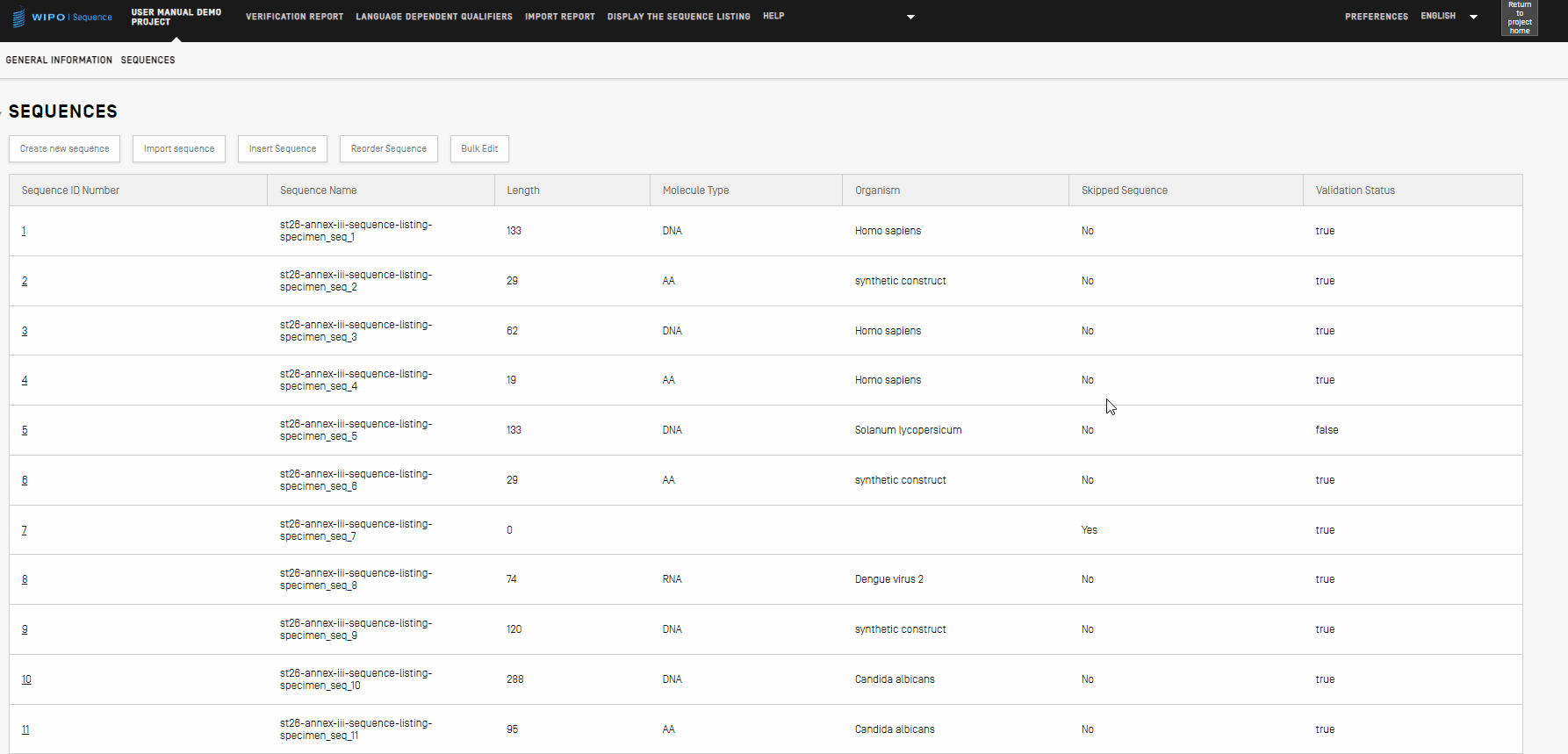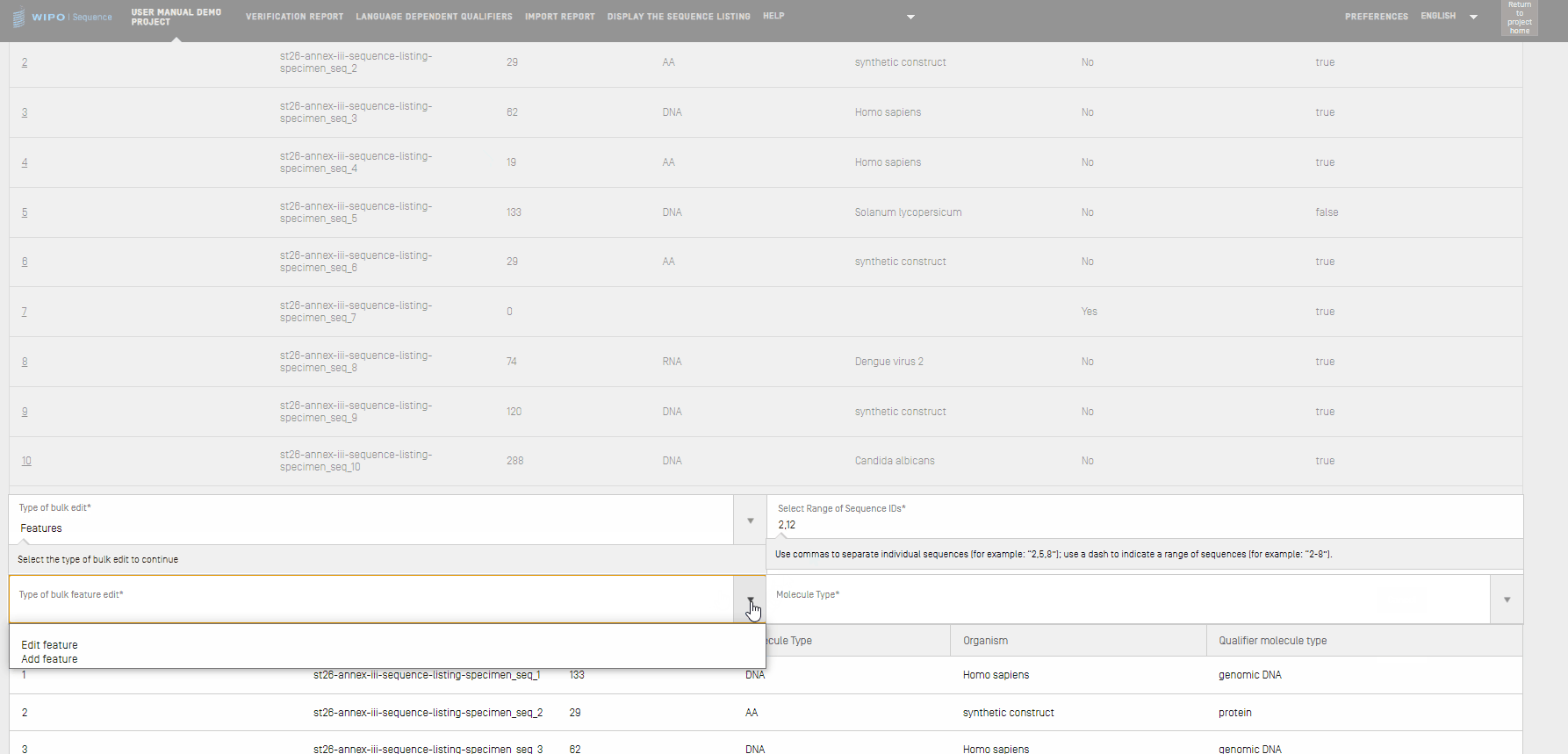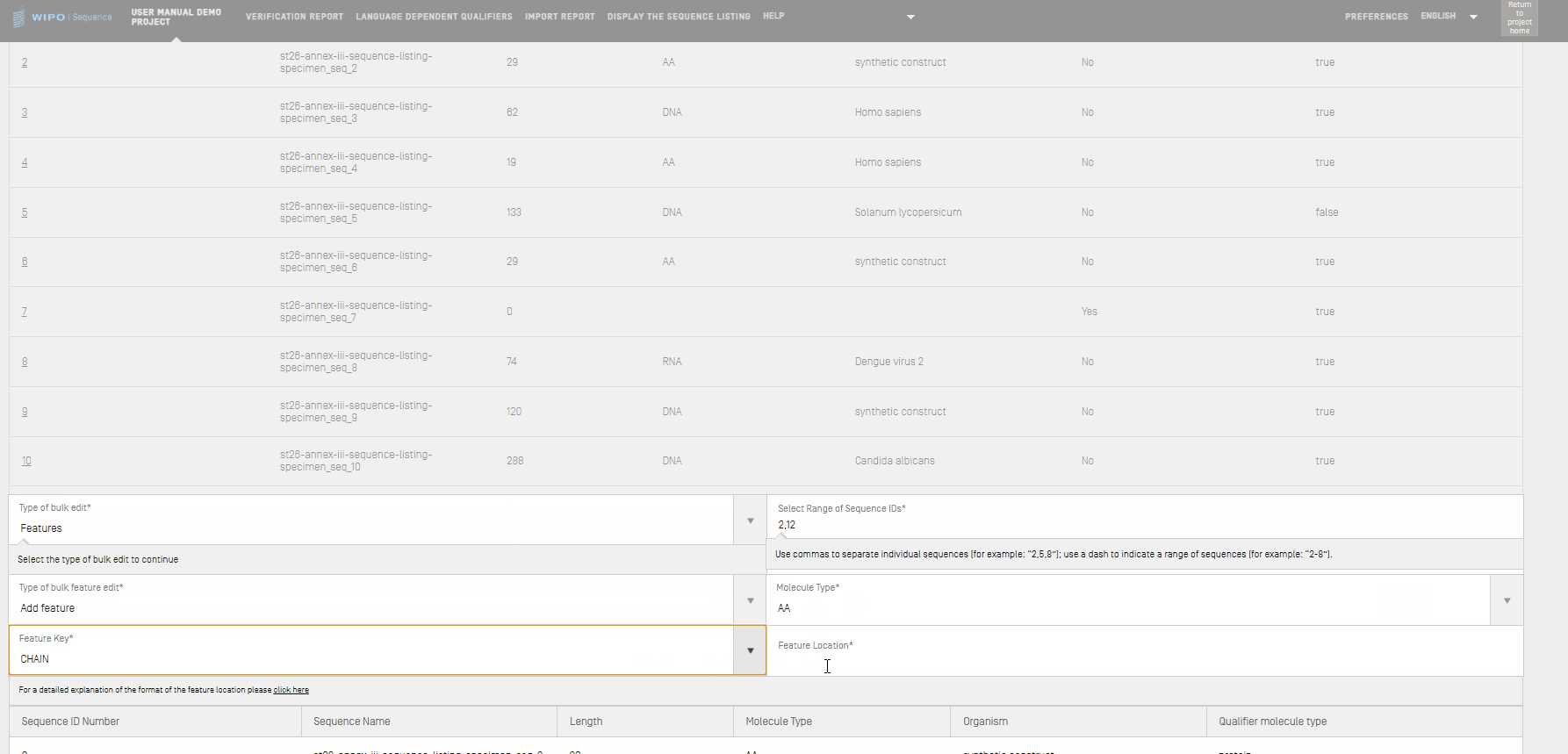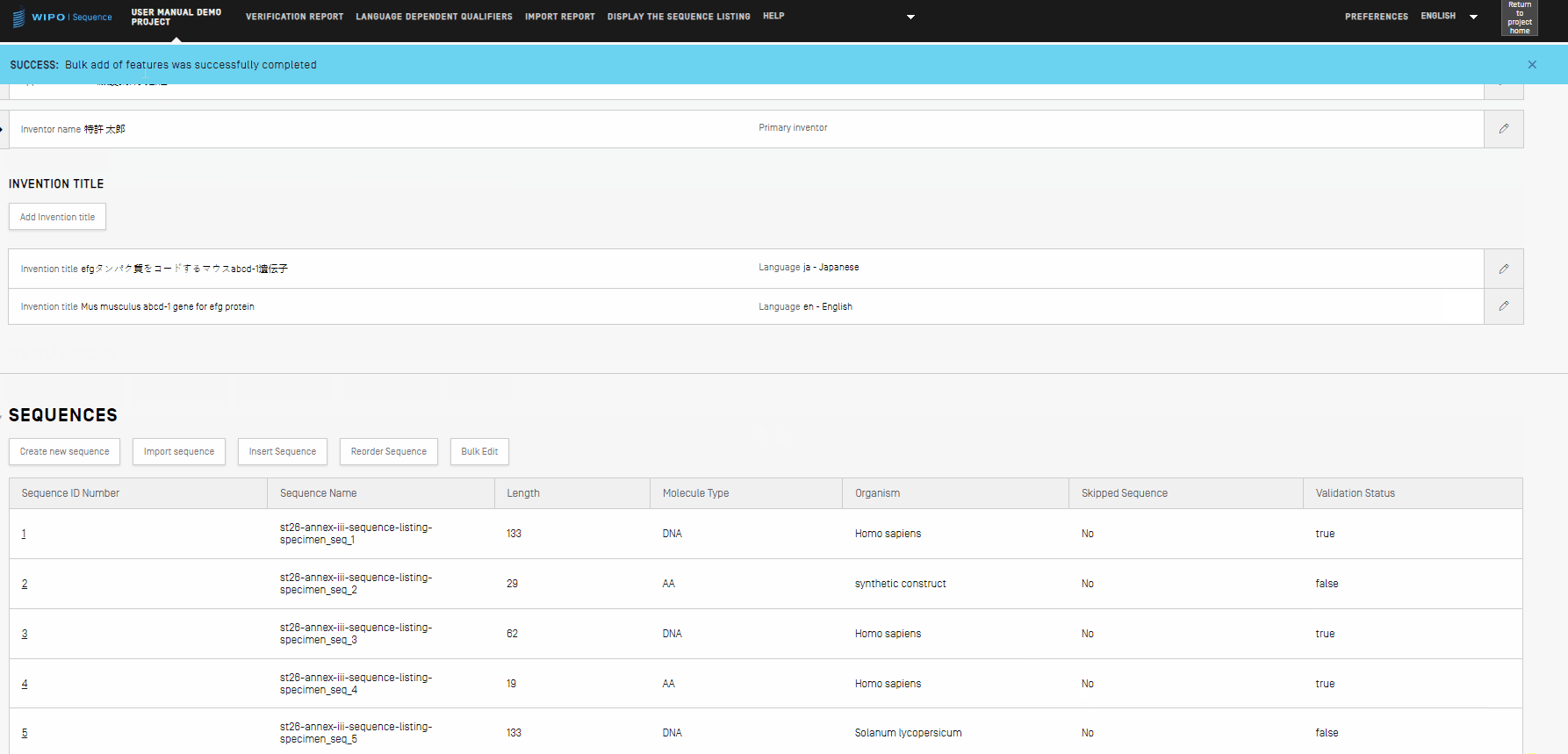
一括編集
どの配列も鉛筆アイコンをクリックして1つずつ編集できますが、複数の配列に対して変更が必要な場合は、一括編集機能を使用することもできます。個々の配列に対し個別に編集を行うことは可能であるものの、配列の数が多いプロジェクトでは非現実的です。この方法で編集できるフィールドは複数あり、詳細は以下で説明します。
- [Bulk Edit(一括編集)]ボタンをクリックして開始します。
- ドロップダウンリストから[Type of bulk edit(一括編集の種類)]を選択します。
- [Qualifier molecule type(Qualifier分子型)]を選択すると、一括編集を適用する核酸配列の種類を選択するよう、システムから求められます。システムは、生物=「合成配列」の配列の「mol_type」のqualifierが「他のDNA」または「他のRNA」でなければならない点、及びこれらの値を変更するとプロジェクト検証時にエラーが発生する点を、ユーザーに警告します。指定された特性を持つ一括編集用の配列のプレビューが表示されます。 編集中、システムは、核酸配列のみ「mol_type」のqualifierの値が編集可能であることをユーザーに通知します(アミノ酸配列に対する同じ値が、システムによって自動的に「タンパク質」に設定されるからです)。
- [Organism(生物)]を選択した場合は、編集する配列IDの範囲を入力する必要があります。次に、生物の値を[synthetic construct(合成コンストラクト)]に変更することを選択した場合、システムは、「mol_type」のqualifierが、配列の分子型に応じて自動的に「他のDNA」または「他のRNA」に変更されることをユーザーに通知します。
- featureを選択した場合は、既存のfeatureを編集するか、新しいfeatureを追加するかを指定する必要があります。「分子型」および編集する配列IDの範囲を入力する必要があります。
- CDS feature位置の値を complement(join(1..30,61..90) に変更するなどして、featureの一括編集の種類を edit featureとして選択した場合、まず [Select Range of Sequence IDs(配列IDの範囲を選択)]のテキストボックスに、関連する配列 ID、分子型、関連する feature key とその feature location を入力します。終了するには、[Edit sequences(配列を編集)]を選択します。
- feature locationが1..4の新しいCHAIN featureを追加するなどして、featureの一括編集の種類を add featureとして選択した場合、まず [Select Range of Sequence IDs]のテキストボックスに、関連する配列 ID、分子型、関連する feature key とその feature location を入力します。
- [Bulk skip(一括スキップ)]を選択した場合は、[Select Range of Sequence IDs]のテキストボックスに、スキップする配列IDの範囲を入力する必要があります。
- [Bulk delete(一括削除)]を選択した場合は、[Select Range of Sequence IDs]のボックスに、削除する配列の範囲を入力する必要があります。
- すべての一括編集操作について、[Edit sequences]ボタンをクリックした後、ツールは、青いバナーを表示して、操作が成功したことを通知します。以下に例を示します。
featureデータの入力
WIPO ST.26では、すべての配列には、少なくとも1つのsource featureが関連づけられていなければなりません。各ソースfeatureには、「生物」と「mol_type」の 2 つの必須qualifierが必要です。
Featureテーブルには、feature key、遺伝子配列内のfeatureの位置、および個々の配列featureに関連付けられたqualifierの3つの列があります。
featureの位置は、配列のどのセグメントにfeatureが存在するかを示します。 featureの位置の指定に使用できる形式は、WIPO ST.26で示されており、以下の通りです。
- 単一残基番号:x
- 配列長さを区切る残基番号:x.. y
- 最初に指定された残基番号の前または最後に指定された残基番号より後の残基:<x, >x, <x..y, x.>y, <x..>y
- 隣接する2つのヌクレオチド間の位置:x^y
- 鎖内クロスリンクで結合した残基番号:x..y
位置演算子は、複雑な位置を記述するために使用できます。
- 「join(位置, 位置, ... 位置)」: 位置は結合され(両端に配置され)1つの連続した配列を形成します。
- 「order(位置, 位置, ... 位置)」: 要素は指定された順序で検出されますが、これらの要素の結合の妥当性については何も示されていません。
- 「complement(位置)」:5'から3'の方向または5'から3'の方向に類似する方向で読んだ場合、位置記述子で指定された配列長さに相補的な鎖にfeatureが位置していることを示します。
配列に新しいfeatureを追加するには、選択した配列のFeatureセクションにある[Add feature(featureを追加)]ボタンをクリックします。この段階でqualifierをfeatureに追加することもできます。これについて、次のセクションでより詳細に説明します。
CDS feature
CDS featureの種類は、タンパク質のコード配列を記述するために使用します。CDS featureは、それが属す配列のセグメントのアミノ酸翻訳を任意で含むことができます。これが最小長の要件を満たしていれば、プロジェクト内で別個の配列として現れます。元の配列のCDS feature内には、「protein_id」qualifierで指定される翻訳アミノ酸配列の配列IDへの参照があります。
配列の「CDS」featureを作成する場合、featureの位置によって示されるように配列の翻訳された残基チャンクに対応するqualifier値を使用して「translation」qualifier(デフォルトの「遺伝コード」の値は1 –「標準コード」)をCDS featureに自動的に追加できます。また、関連する「プロテインID」と別個のアミノ酸配列を、プロジェクト詳細ページの上部にある基本情報のチェックボックスをオンにすることにより生成することもできます。ただし、このqualifierは必須ではなく、作成後に削除できます。ユーザーが作成した、関連する翻訳済み配列IDを参照する「翻訳」および「protein_id」のqualifierを手動で作成することもできます。
注:バージョン 2.1.0 からは、[Automatically add a translation qualifier(translation qualifierを自動的に追加する)]のチェックボックスはデフォルトでチェックされています。
CDS feature qualifierを自動的に作成する手順は、次の通りです。
- 特定の配列を表示して、[Add feature(featureを追加)] ボタンをクリックし、feature keyとして「CDS」を選択します。基本情報のautomatically add a translation qualifierチェックボックスがオンになっている場合、CDS featureをヌクレオチド配列に追加すると、translation qualifier、その値、protein_id qualifierおよび関連付けられた別のアミノ酸配列(該当する場合)が自動的に追加されます。
- translation qualifierを手動で作成することも選択できます。
- featureとそれに関連するqualifierの編集を終了したら、ユーザーは[Create Feature(featureを作成)] ボタンをクリックして保存してください。それにより得られるCDS featureが、配列に関連付けられて示されます。
translation qualifier の値が最小長の要件を満たしている場合、ツールは次の属性を持つプロジェクトの新しい配列を作成します。
- 配列ID番号=配列ID番号で次に使用可能な値
- 長さ=翻訳された配列の長さ
- 配列の名称 = translation qualifierの[Sequence Name(配列の名称)]フィールドで指定された値。名称を指定しなかった場合、デフォルトの配列名(「Seq_#」) が指定されます。
- 分子型=「AA」
- 生物の名称=元の配列に指定された値と同じ値
- Qualifier分子型 =「タンパク質」
- 配列残基 =元の配列の翻訳された値
注:翻訳配列の作成について、別個の翻訳配列は、少なくとも 4 つの具体的に定義された残基が定義されている場合にのみ作成されます (たとえば、「AXTG」は 3 文字としてカウントされます)。「translation」 qualifierを変更する際、qualifierの値が含む具体的に定義された残基の数が4未満であると、「protein_id」qualifierと同様に、関連付けられた配列の翻訳が削除されます。
疑似または偽遺伝子qualifierを含める場合のCDS featureに関するアドバイス:
CDS featureに疑似または偽遺伝子qualifierを追加するときは、自動翻訳がオフになっていることを確認してください。CDS featureに疑似または偽遺伝子qualifierが追加される時に自動翻訳がオフになっていない場合、CDS featureが更新されると、translation qualifierが自動的に追加されます。このエラーを修正するには、プロジェクトの自動翻訳をオフにしてから、CDS featureを開き、翻訳および「protein_id」のqualifierを削除してから、featureを更新します。
translation qualifierを自動で作成したい場合は、qualifierの編集パネルから翻訳テーブルの値と配列の名称を設定できます。featureを作成すると、ツールは翻訳を実行し、featureに「protein_id」qualifierを追加し、翻訳値を持つ新規配列を追加します。
featureの位置またはqualifier「transl_table」、「transl_except」、もしくは「codon_start」の値が変更された場合にのみ、翻訳が再実行されます。この場合、関連付けられた配列は更新されます。
注:翻訳値が変更されると、関連付けられた配列は自動的にその値を更新します。しかし、関連付けられたヌクレオチド配列が変更された場合、translation qualifierの値は変化しません。作成後に「protein_id」qualifierを変更すると、関連付けられた配列は元の配列との関連付けを失います。
終止コドンの使用に関するアドバイス:
一般に、終止コドンはCDS featureの最後にのみ存在し、コードされたアミノ酸配列のエンド・ポイントを示します。終止コドンが特定のアミノ酸に変換されることを示す「transl_except」の qualifierがない限り、CDS featureの途中に存在するべきではありません。
CDS featureの途中(下記では黄色で強調表示)に終止コドンが存在し、終止コドンが特定のアミノ酸に翻訳されることを示す「transl_except」の qualifierが存在しない場合、ツールはその位置で翻訳を停止し、翻訳が作成されないことをユーザーに知らせる赤いバナーが表示されます。
コード配列に問題があることをユーザーに警告するために、検証レポートにエラーが表示されます。
qualifierデータの入力
qualifierは、feature keyとfeature locationによって表される情報に加えて、featureに関する情報を提供するために使用されます。qualifierによって表される様々な種類の情報に対応するために、3 種類の値形式があります。
- フリーテキスト
- 統制語彙または列挙値(数値や日付など)
- 配列
featureのqualifierを表示するには、まず、該当する配列のfeatureテーブルから該当するfeatureを選択してください。その上で、鉛筆アイコンをクリックしてオーバーレイを開きます。
既存のqualifierは、各行の右側にある鉛筆アイコンをクリックして編集できます。または、[Add qualifier(qualifierを追加)] ボタンをクリックして、現在選択されているfeatureに新しいqualifierを追加できます。
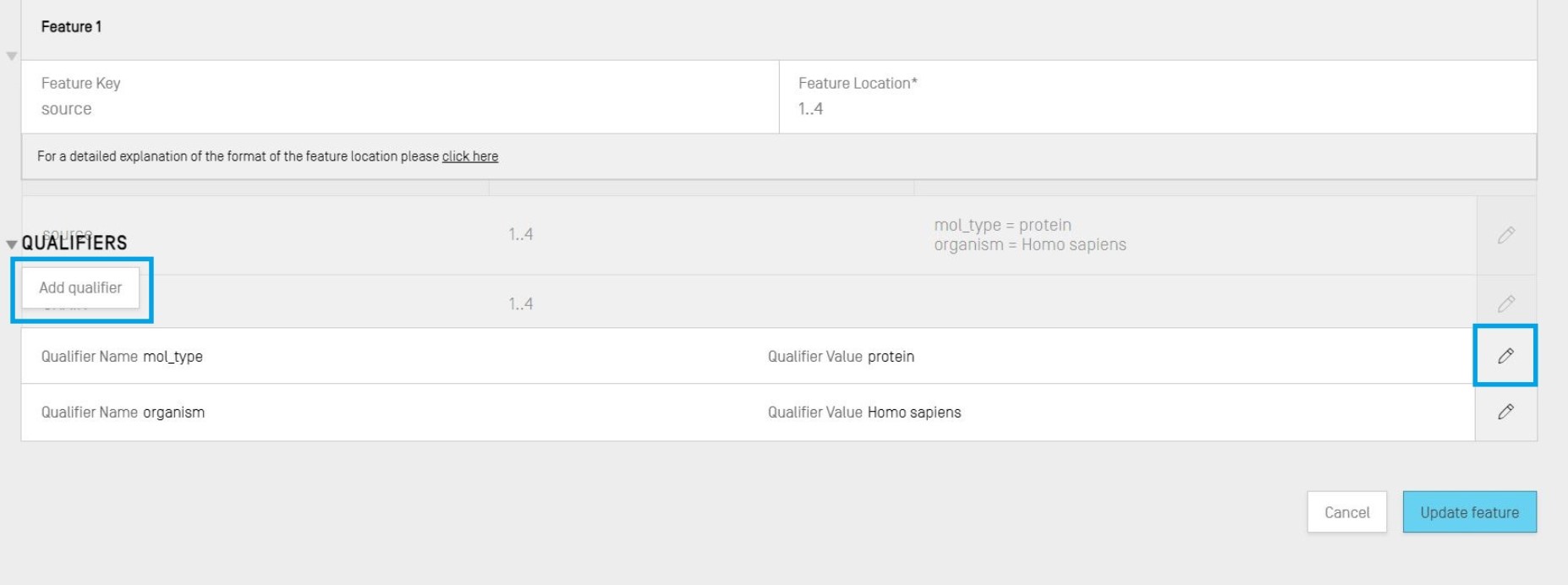
qualifierを編集または追加すると、[Qualifier name(Qualifier名)](ドロップダウンリストから選択)と [Qualifier value(Qualifierの値)] の2つのフィールドがユーザーに表示されます。
Qualifierの値のフィールドの動作は、qualifierのタイプによって異なります。
- 事前に定義された値を持つqualifier:値のフィールドは、qualifierの定義済みの値から1つを選択できるドロップダウンフィールドです。
- フリーテキストを含むqualifier:値のフィールドはフリーテキストフィールドです。Qualifier名および英語の値のみを有するQualifierの値に加えて、言語コード(「ru」など)の入力と対応する言語の値のNon English Qualifier Value(英語以外のQualifierの値)への入力をできるように、2つの追加のフィールドが表示されます。言語コードのフィールドには、プロジェクト詳細情報の「英語以外のフリーテキスト言語コード」と同じ値が割り当てられます。手入力により、またはXLIFFファイルから適切な関連言語をインポートすることにより、選択した各言語に対して一連の非英語の値を入力できます。
- 事前に定義された形式のqualifier:値フィールドはフリーテキストフィールドですが、入力した値はWIPO ST.26 Annex I, Section 6に記載されている特定の規則と一致することを確認するために検証されます。
- 使用可能な値のないqualifier:qualifierの値フィールドは編集できません。
完了したら、青色の [Create Qualifier(Qualifierを作成)] ボタンをクリックして新しく作成したqualifierを追加するか、または [Save(保存)] をクリックして既存のqualifierに加えた変更を保存してください。最後のステップでは、qualifierの追加/変更が完了したら、オーバーレイの下部にある [Update feature(featureを更新)]ボタンをクリックしてください。
個人および法人ビュー
このビューは、ローカルに保存されているすべての個人と法人を管理します。
個人/法人を作成
新規個人または法人を作成するには、個人および法人ビューから開始してください。以下のように、ビューの先頭にある [CREATE NEW PERSON OR ORGANIZATION(新規個人または法人を作成)] リンクをクリックしてください。
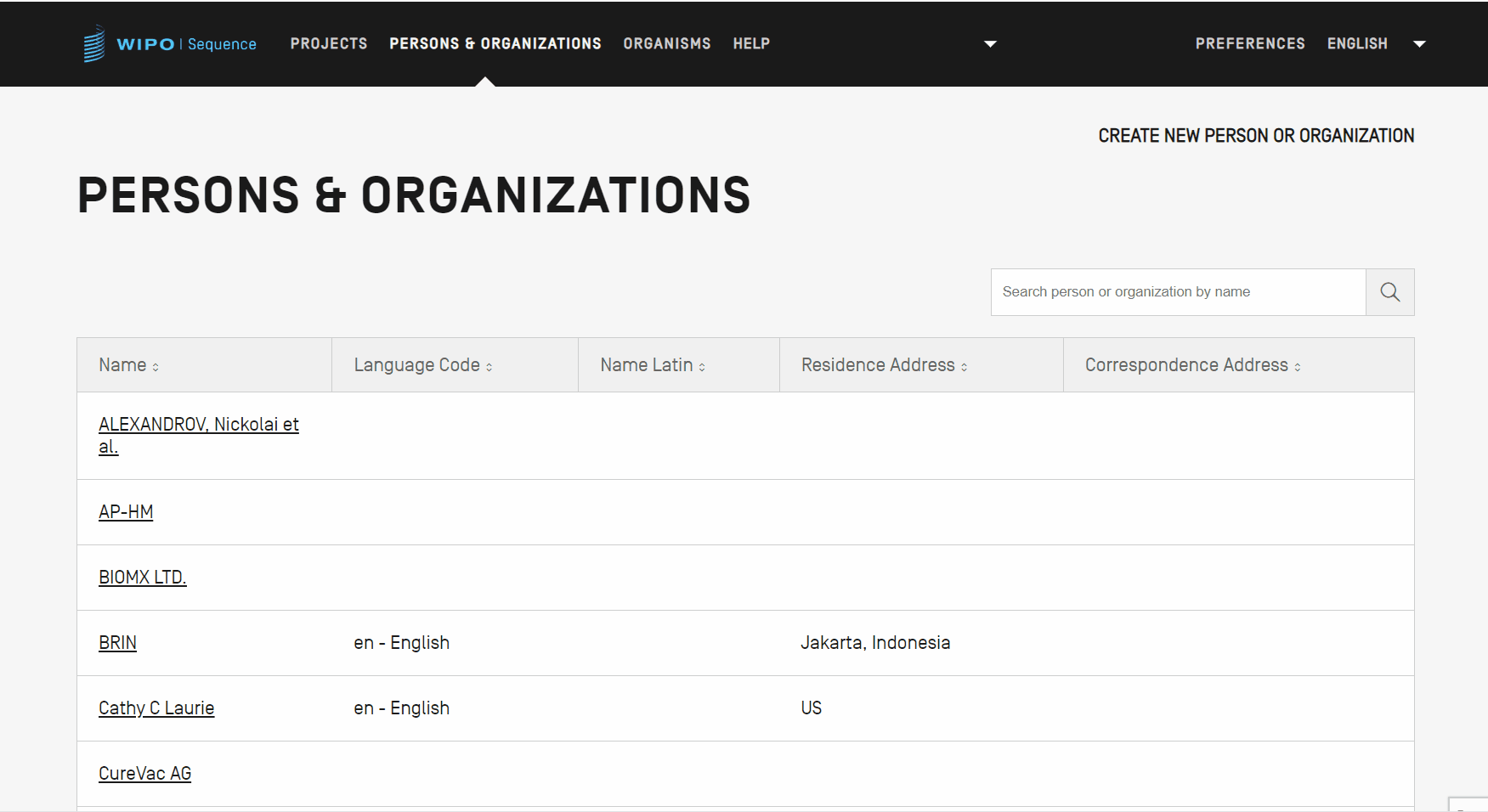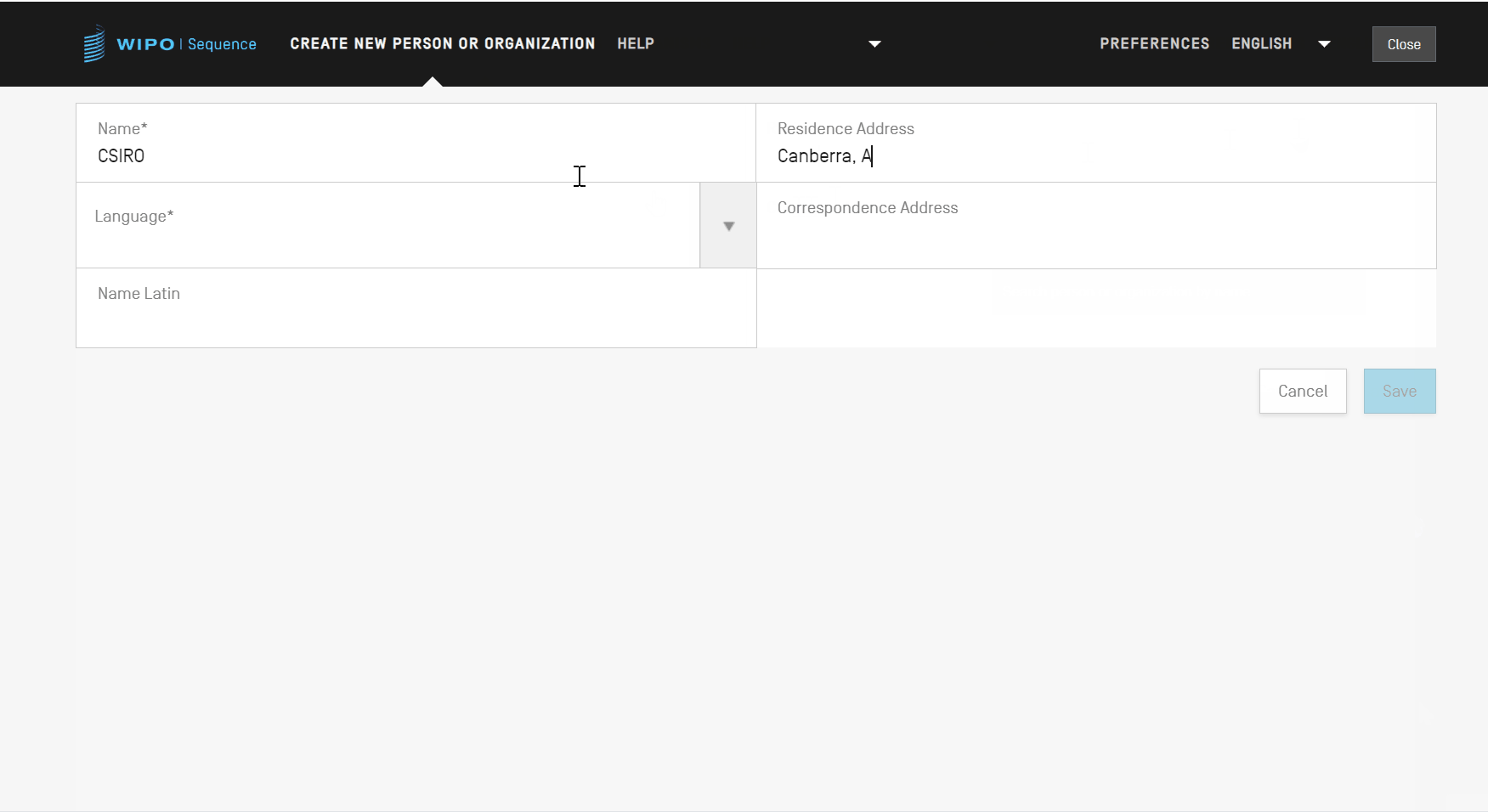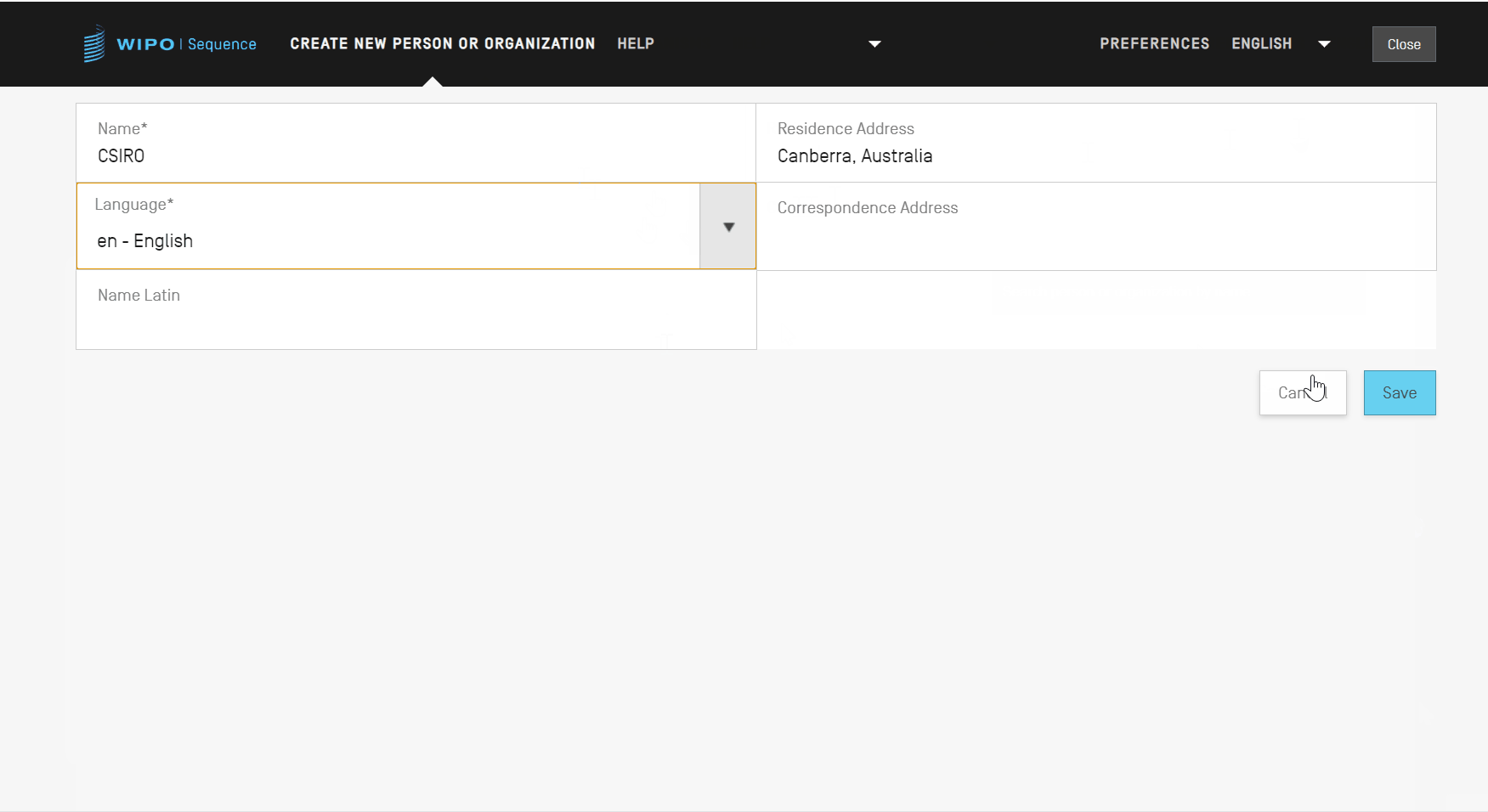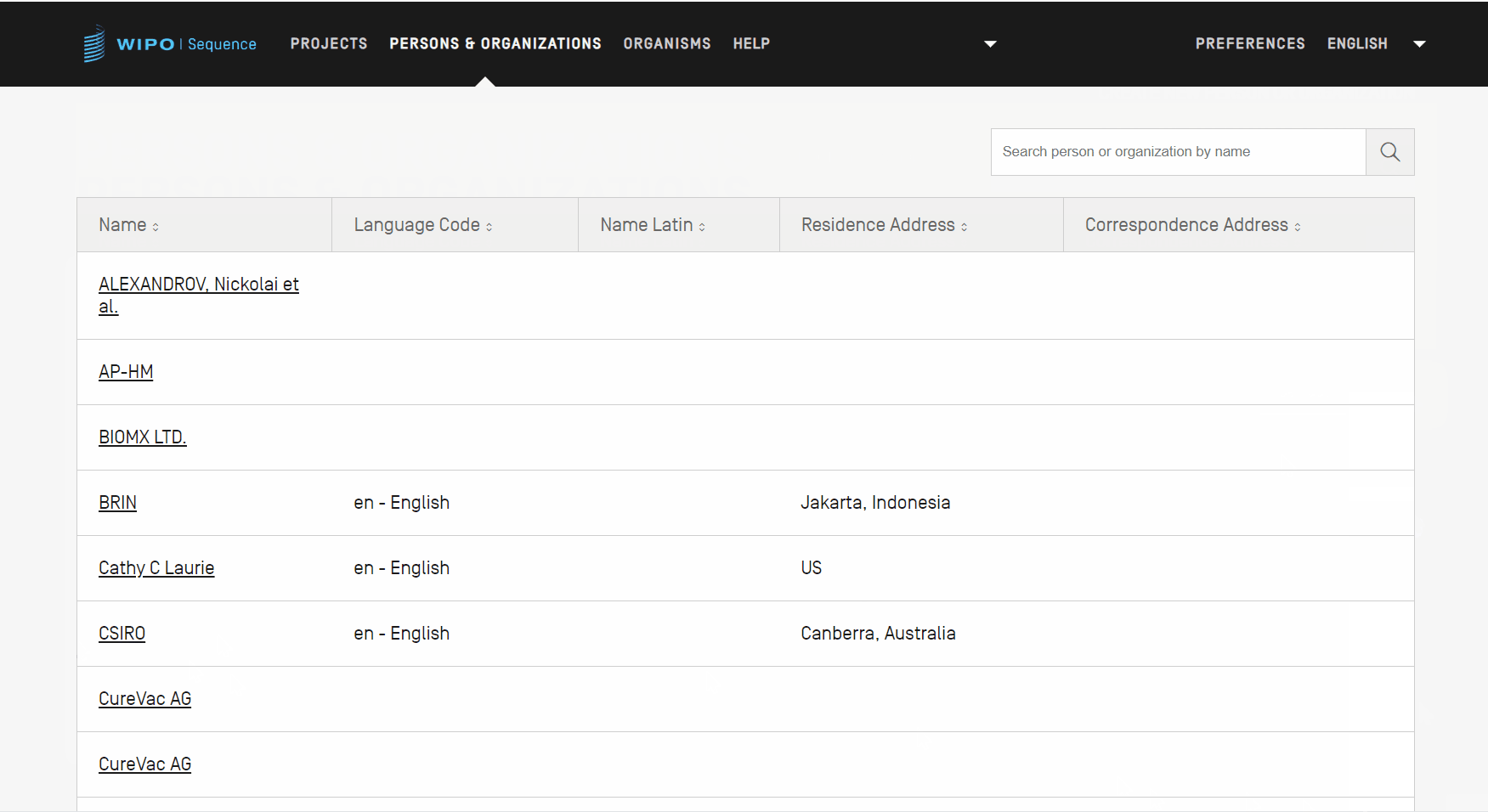
新しいビューでは、ユーザーは少なくとも新規個人/法人の詳細に対応する必須のフィールド(「*」で示す)を入力してください。出願人/発明者にとって、これは名前(ラテン文字で提供されている場合)と言語のみです。
個人または法人の名称がラテン文字でない場合は、名称のラテン語表記を「ラテン語名」フィールドに入力する必要があります。この情報が入力されていない場合、ST.26配列表が検証あるいは作成される際にプロジェクトが検証されません。
2.4 ユーザーの生物ビュー
ユーザーの生物を作成、編集、インポート、エクスポート、または削除するには、生物ビューから開始してください。
ユーザーの生物の作成
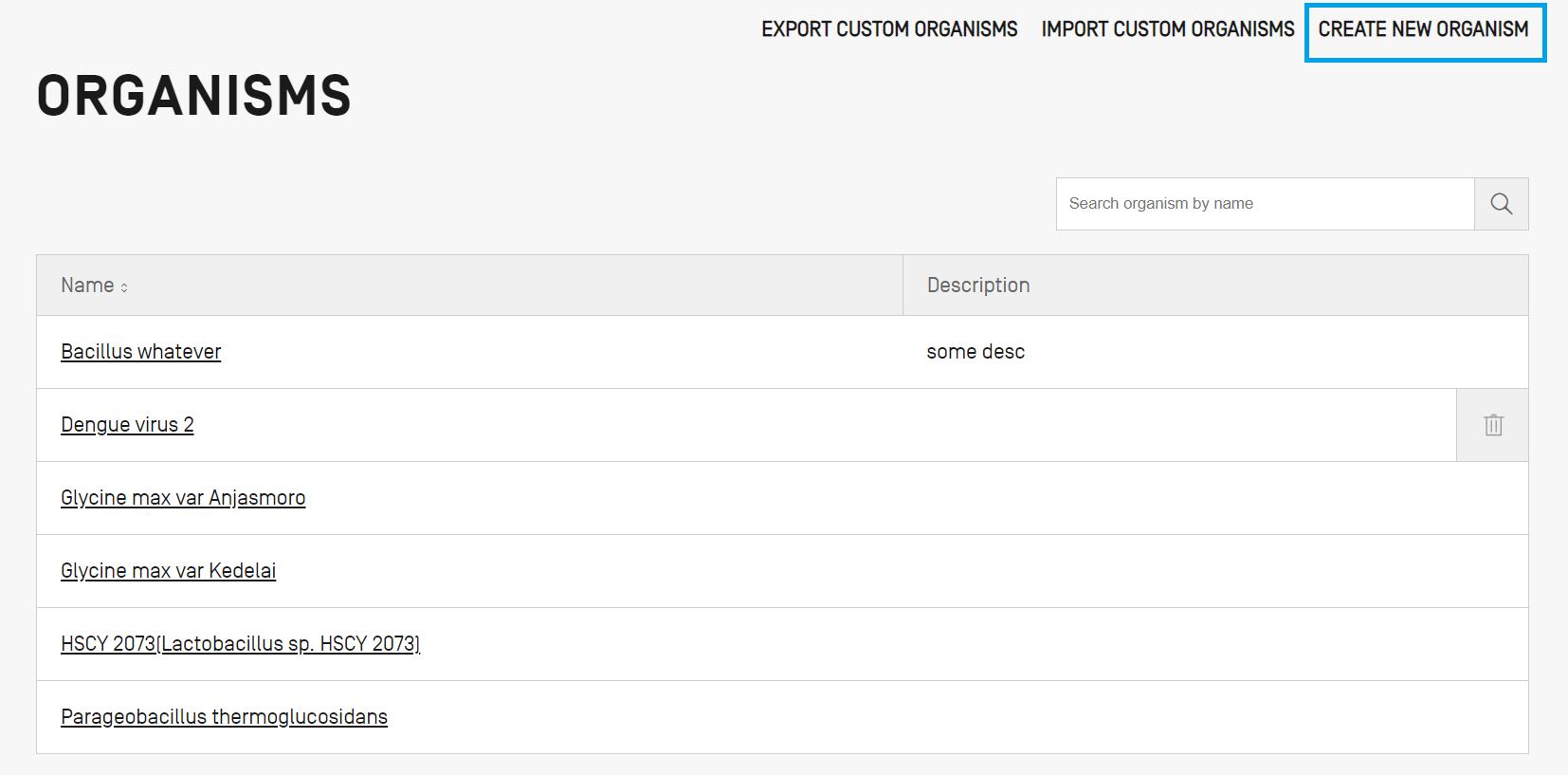
新しいユーザーの生物を作成するには、ビューの先頭にある [CREATE NEW ORGANISM(新規生物を作成)] リンクをクリックします。次の画面で、新規生物の名前を入力し、[Save(保存)] をクリックします。このユーザーの生物の説明が必要な場合は、以下で例示するようにオプションで追加できます。詳細を編集するには、生物の名前をクリックします。
ユーザーの生物をエクスポート
ツールに保存されているすべてのユーザーの生物および説明は、エクスポートしてテキストファイルに保存し、ツール外で修正したり、後でインポートすることができます。このリストをエクスポートするには、以下で強調表示されているように [EXPORT CUSTOM ORGANISMS(ユーザーの生物のエクスポート)] を選択することから始めます。

次に、ダイアログボックスが開き、ファイル名と所望のファイルの場所を選択できます。

エクスポートされたファイルは、編集してツールにインポートすることが可能な生物の名前と説明の両方を含むtxtファイルです。一例をダウンロード。
ユーザーの生物をインポート
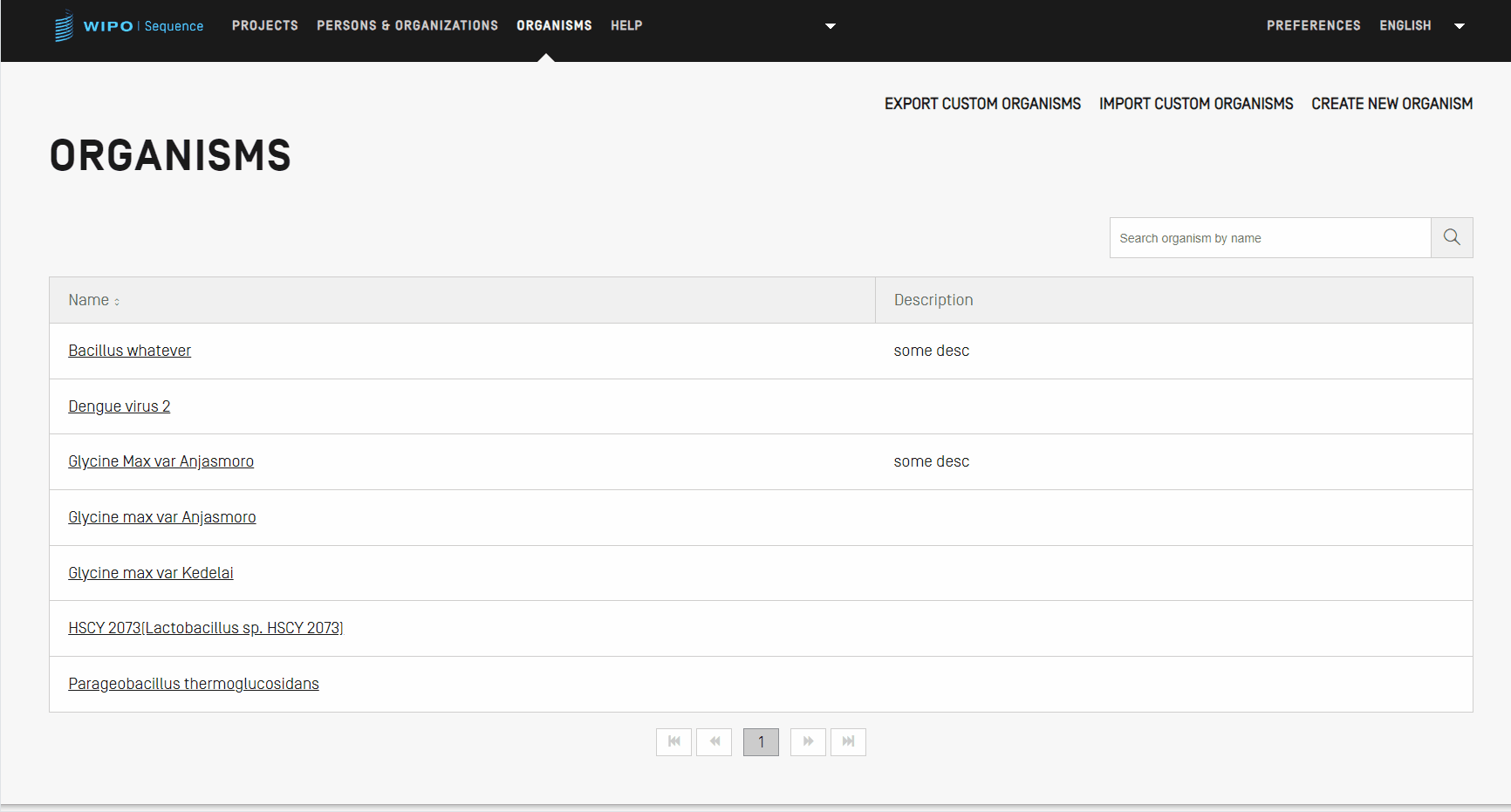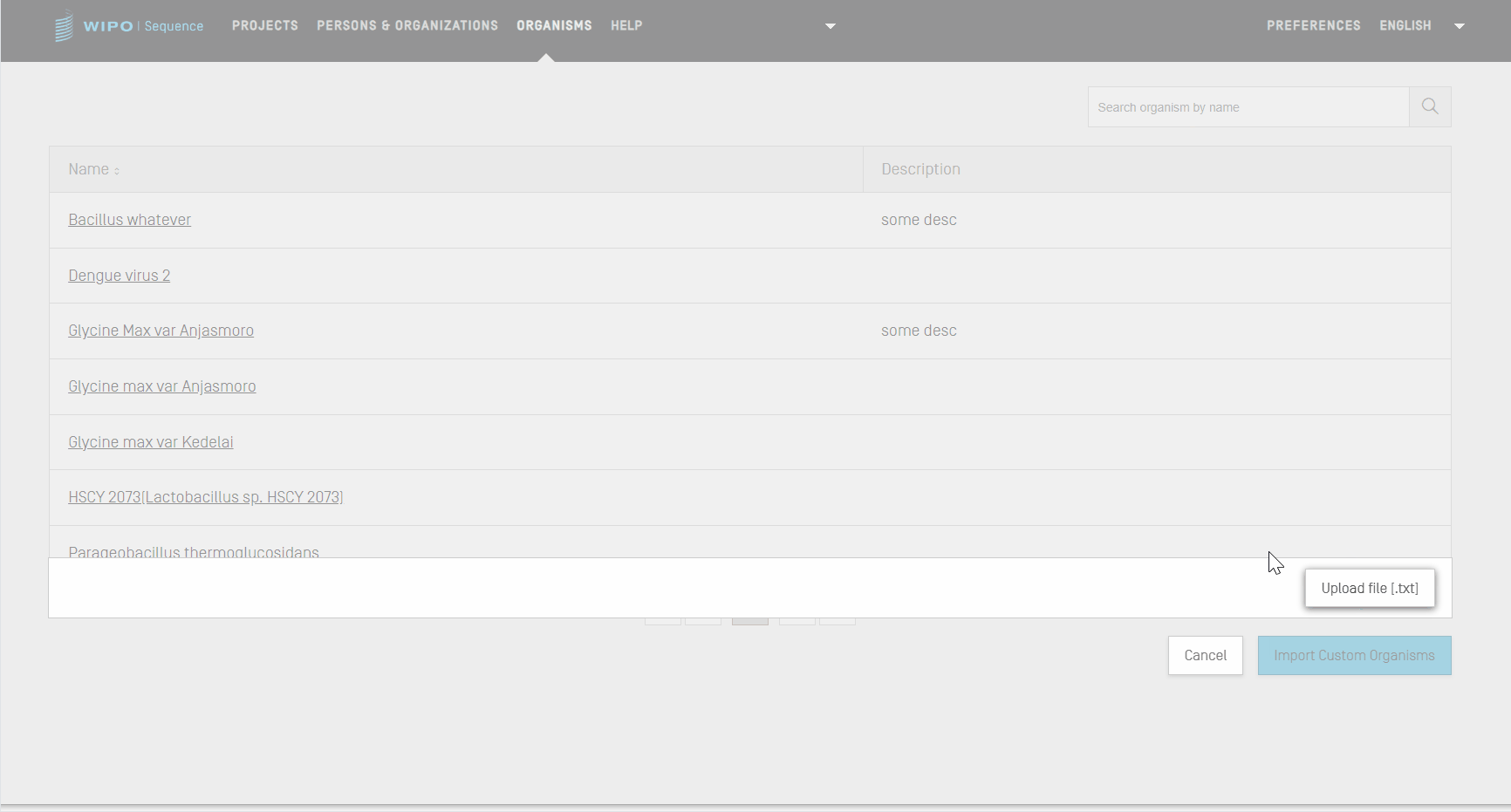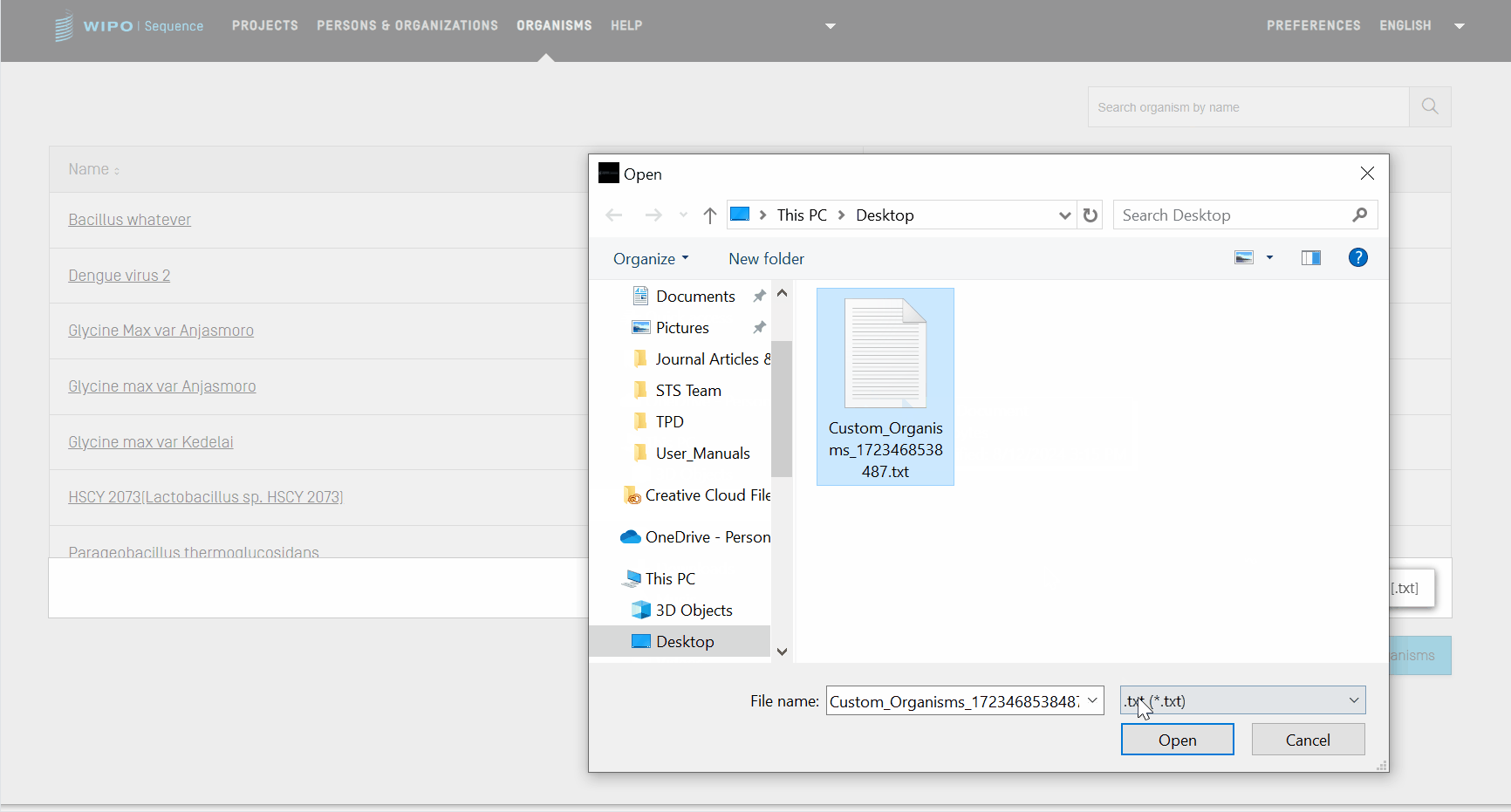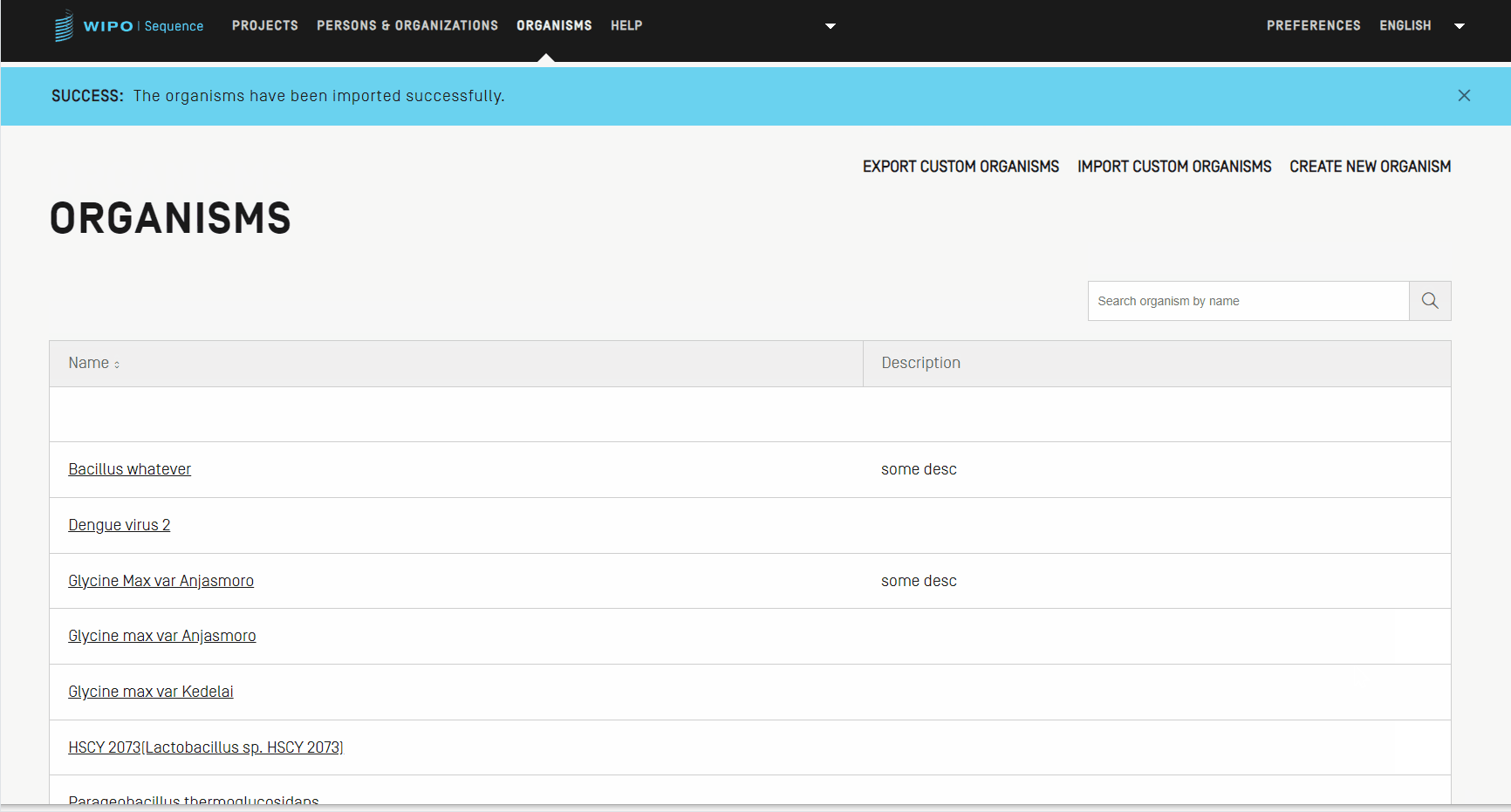
まず、ユーザーの生物のリストをインポートするには、ビューの先頭にある [IMPORT CUSTOM ORGANISMS(ユーザーの生物をインポート)] リンクをクリックしてください。これにより、ユーザーの生物のサマリーテーブルの下にオーバーレイが開きます。
- [Upload file [.txt](ファイル [.txt] をアップロード)] ボタンをクリックします。
- ダイアログボックス内から、ユーザーの生物名を含むファイルを選択します。
- 最後に、青色の [Import Custom Organisms(ユーザーの生物をインポート)] ボタンをクリックします。
注:インポートされるファイルは、ユーザーの生物名のリストがプレーンテキスト(UTF-8)であり、各項目が改行されたテキストファイル(*.txt)です。
2.5 システム環境設定ビュー
システム環境設定ビューでは、WIPO Sequenceのいくつかの構成パラメータを変更できます。これらのパラメーターは、ツールによって作成または編集されたすべてのプロジェクトに適用されます。
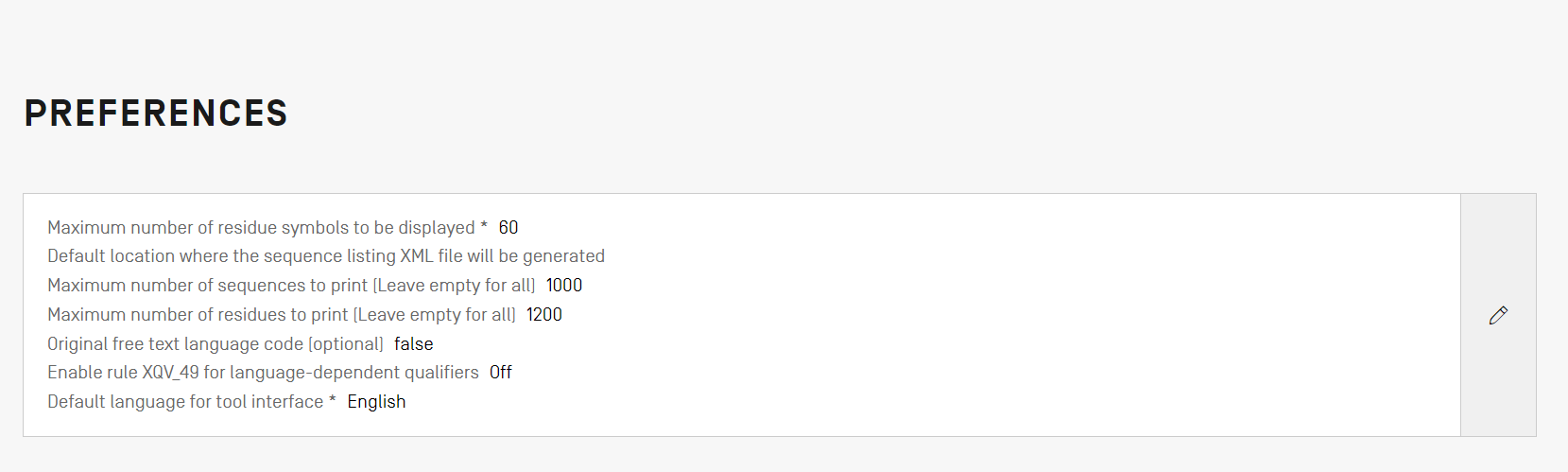
システム環境設定を変更するには、上に示した鉛筆アイコンをクリックして、編集パネルを開いてください。
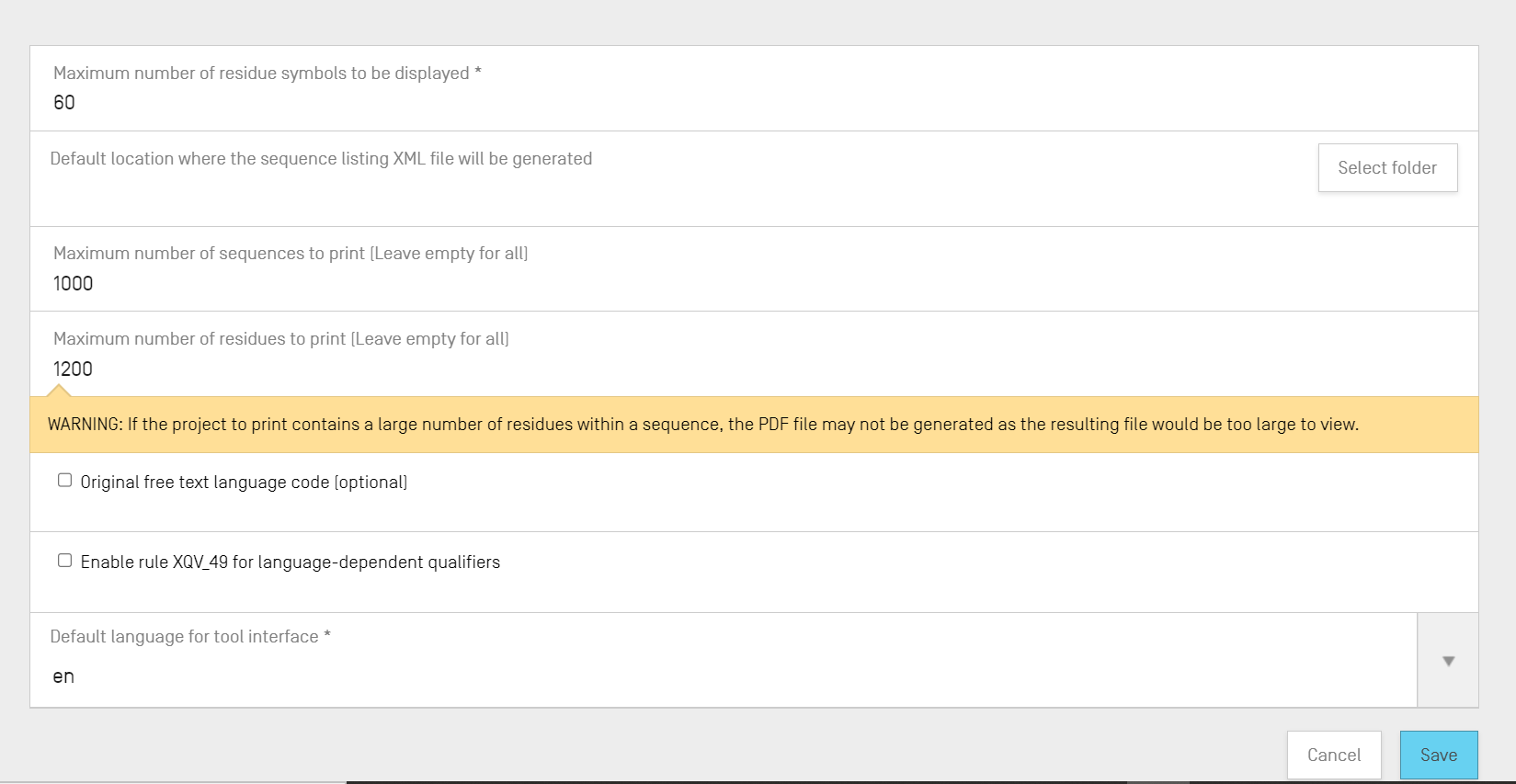
このビューから変更できる構成項目のリスト(順に沿って記載)は次の通りです。
- 表示される残基記号の最大数。このパラメータは、配列を表示する際、行ごとに表示される残基の数を設定します。デフォルトでは60です。
- ST.26配列表ファイル(.xml)が作成されるデフォルト位置。これを入力する必要はありません。
- 印刷する配列の最大数(すべての場合は空欄とする):デフォルトは1000。
- 印刷する残基の最大数(すべての場合は空欄とする):デフォルトは1200。
- 元のフリーテキスト言語コード:このチェックボックスがオンにされている場合、元のフリーテキスト言語コードが指定されていなければ、検証中に警告があります。デフォルトでは、チェックされていません。
- XQV_49を有効にする:このチェックボックスがオンにされている場合、言語依存フリーテキストqualifierに英語の値が指定されていなければ、警告があります。デフォルトでは、オフになっています。
- デフォルトのインターフェイス言語: これは、WIPO Sequenceの起動時にインターフェイスが表示される言語です。デフォルトでは、英語です。
注:3番目および4番目の項目は、プロジェクトをPDFで印刷することに関連しています。非常に大きな配列表の場合、生成されるPDFが数千ページになり、表示できないことがあります。
3. ファイル形式
WIPO Standard ST.25
WIPO Standard ST.25ファイルの形式の詳細については、Standardを参照してください。
例は、StandardのAnnex IIIとして提供されています。
RAW
この形式では、1つの配列のみ記述できます。 遺伝暗号はその基本形で書かれており、追加情報はありません。 インポート時には、分子型、feature、名称をツールから配列に追加してください。
例:
aggatatagatagtatatgatagtatgatatgatgatgtatgtatagtgtagttatga
マルチシーケンス
マルチシーケンス形式は、1つまたは複数の配列をそれらの名称、分子型、生物の名称とともに記述することができ、「PatentIn」を使用したインポートに使用可能なフォーマットの1つです。空白でないテキストの最初の行はヘッダーであり、次の構成要素から構成されています。
<SequenceName; SequenceType; OrganismName>
ヘッダーの情報は、WIPO Sequenceによって次のように解釈されます。
| Header entry(ヘッダーエントリ) | Allowable Input(許容される入力) | Interpreted as(以下のように解釈する) |
|---|---|---|
| 配列名 | 配列の名前 (フリーテキスト) | WIPOプロジェクトファイル内の配列の名前(XMLファイルの一部ではありません) |
| 配列型 | 以下のうちいずれか:
|
mol_type 注:Organism の入力によっては、DNA と RNA のmol_typeをさらに定義する必要があります (ST.26、 paragraphs 75-84) |
| 生物名 | 生物名(フリーテキスト) | ソース/生物 注:インプットが合成コンストラクトである場合、DNAおよびRNAのmol_typeは自動的に「他のDNA」または「他のRNA」として識別されます(ST.26、paragraph 84(a))。 |
配列データは、ヘッダーの後の行から始まります。新しい配列は、前の配列の配列情報の後ろでファイル内の新しい行に記述されます。ある配列の末尾と次のヘッダーの開始との間には、1 つ以上の空行がある場合があります。アミノ酸配列は1文字のコードである必要があります(Annex I、Section 3、Table 3)。許容されるヌクレオチド記号は、Annex I、Section 1、Table 1のものです。なお、RNA配列の「u」は、ST.26のparagraph 14および19で要求されているように自動的に「t」に変換されず、インポート後に手動による操作が必要になります。RNA配列を含むファイルのマルチシーケンスインポートを実行する前に、「u」を「t」に変換することをお勧めします。
以下は、マルチシーケンス形式で定義された3つの配列のセットの例です。
例:
<First Sequence; RNA; Albies alba> uuuucuuauuguuucuccuacugcuuaucauaaugauugucguaguggcuuccucaucgucucccccaccgccuaccacaacgacugccgcagcggauuacuaauaguaucaccaacagcauaacaaaaagaaugacgaagaggguugcugauggugucgccgacggcguagcagaaggaguggcggagggg
<Second Sequence; DNA; synthetic construct> attgacgtcagtgacgcggtactgacgtcagctgcagtactgacgtaccaaccacgtggtgagctctcgacatgcaactgactcgtcgctattgacgtcagtgacgcggtactgacgtcagctgcagtactgacgtaccaaccacgtggtgagctctcgacatgcaactgactcgtcgctcagt
<Third Sequence; AA; Mus musculus>
SPPGKPQGPPPQGGNQPQGPPPPPGKPQGPPPQGGNRPQGPPPPGKPQGPPPQGDKSRSPR
FASTA
この形式には残基と説明が含まれています。インポート時、説明をnote qualifierとして保存することを選択できます。
例:
AJ011880.1 人工オリゴヌクレオチド配列SSRプライマー (CAC13R)
CTCAACAATCTGAAGCATCG
4. トラブルシューティング
WIPO Sequenceには、トップメニューからアクセスできる「ヘルプ」機能があります。
- ヘルプオプションで誘導する情報は次のものを提供します:
- ユーザーマニュアルへのリンク
- WIPO ST.26 Knowledge Base へのリンク
- WIPO Sequenceサポートチームのお問い合わせフォームへのリンク
- 最新版WIPO Standard ST.26へのリンク
- WIPO シーケンス・デスクトップツールのバージョンに関する基本情報
よくある問題と解決
インポートレポートまたは検証レポートは、プロジェクトに複数のqualifier ID が含まれていることを示しています。
解決: 重複するqualifier ID を含むfeatureを削除します。同じ場所でfeatureを再作成しますが、保存する前にqualifierを追加しないでください 。次に、featureを編集して、関連するqualifierを追加し、featureを更新します。
Linuxディストリビューションで推奨されるファイル名が正しくありません。
Linux ディストリビューションの使用時に発生する問題が知られています:デフォルトでのファイル名の提案時に余分な '\' が表示されます。この問題を解決するため、保存する前に余分な '\' を手動で削除してください。
他の解答については、WIPO Sequenceのナレッジベースを参照してください
配列表をHTML形式で表示しようとするとエラーが表示されます
XML 形式で生成された配列表のサイズが100 Mbより大きい場合は、配列表を HTML 形式で表示する代わりに、配列表が HTML 形式でレンダリングするには大きすぎることを示すエラーが表示されます。
その他の未解決の質問については、 お問い合わせください。